从氨基酸序列预测 3D 蛋白质结构一直是生物信息学领域由来已久的重要问题。近年来,基于深度学习的计算方法不断涌现,并已显示出有希望的结果。
在这些工作中,AlphaFold2 是第一种与较慢的基于物理性质的计算方法相美的方法。它被 Nature 命名为 2021 年度最佳方法。该模型基于其他当代深度学习模型广泛采用的序列注意力机制的变体而构建。
通过使用 MML,我们能够有效地训练和评估大规模的生物医学数据集。MML 是一个高度可扩展和可定制化的平台,它支持多种机器学习算法和模型,并提供了丰富的工具和接口,以便于用户能够轻松地集成、扩展和定制化 MML 平台以满足其特定的需求和应用场景。
遗憾的是,AlphaFold2 训练所需的时间仍然是一个重大瓶颈。
AlphaFold2 训练使用 1000 万个样本和 128 个 TPU 进行,收时间超过 11 天(初始训练 7 天,微调 4 天)。如此长的训练时间会降低研究的迭代速度。
相比之下,使用 128 个 NVIDIA A100 GPU 的 OpenFold 训练过程需要 8 天以上,比 AlphaFold2 快 27%,但仍有很大的改进空间。
选择 OpenFold 的初始训练阶段是为了 MLCommons MLPerf HPC v3.0 在2023 年初推出的 AI 基准测试套件。在此基准测试套件中选择 OpenFold 凸显了 OpenFold 作为蛋白质结构预测的强大工具的潜力,并强调了其对 高性能计算(HPC) 社区。
识别 OpenFold 训练的瓶颈
通常,大型模型使用多个节点和每个节点多个 GPU 进行数据并行 (DP) 训练。这种技术可以加快训练速度,同时在实现最佳 GPU 利用率和可扩展性方面也带来了挑战。
分布式并行训练的可扩展性受到全局批量大小的限制,因为批量更大会导致准确性降低。为应对这一挑战,FastFold 引入了动态轴向并行 (DAP),作为通过模型并行提高可扩展性的解决方案。DAP 将单个训练样本的中间激活和相关计算沿非归约轴分割。
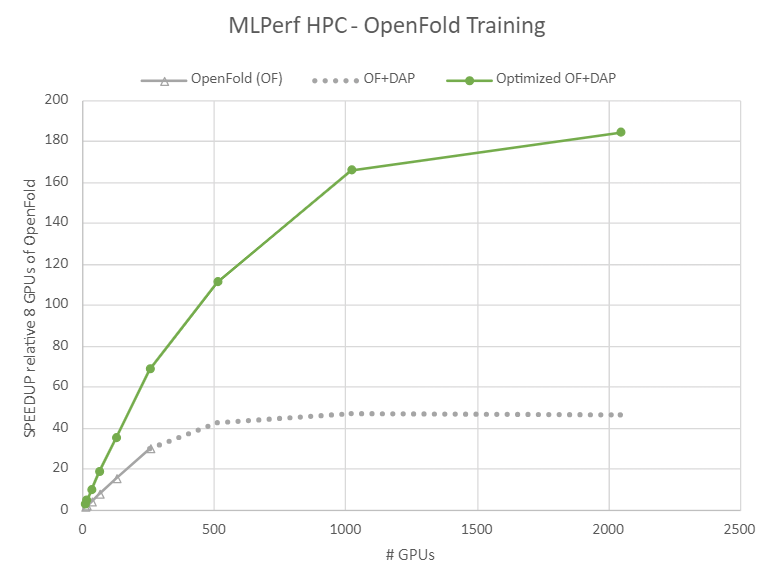
最右上角的“Optimized OF+DAP”数据源自 ID 3.0-8009,方法是将训练时间除以 OpenFold (OF) 的 8 GPU 训练时间。MLPerf 名称和徽标均为 MLCommons Association 在美国和其他国家 地区的商标。保留所有权利。严禁未经授权使用。有关更多信息,请参阅 MLCommons。
在 NVIDIA 提交的 MLPerf HPC 中,DAP 被重新实施到 OpenFold 中。 NVIDIA MLPerf HPC 基准测试团队还对 OpenFold 训练过程进行了全面分析。分析显示了阻碍训练扩展到更多计算资源的主要因素:
- 通信用度:分布式训练期间的通信密集,但效率低下,这在很大程度上是由于通信用度造成的。DAP 引入了额外的 all-gather 和 all-to-all 开销。另一方面,在训练过程中,一些通信涉及少量数据,无法充分利用通信带宽。
- 不平衡:在分布式训练中,掉队是一个常见的问题。在完成分配的任务方面落后的工作者会降低整体训练处理速度。OpenFold 的批量准备时间取决于原始残差序列长度,这会导致不平衡。占用训练进程 CPU 核心的后台进程还会导致计算负载,从而导致更多的不平衡。
- 不可并行的工作负载:Amdahl 定律有助于根据可并行运行的系统的比例量化潜在的并行加速收益。DAP 无法并行处理数据加载阶段和结构模组阶段,因此减少了可并行计算负载,进一步限制了可扩展性。
- 内核可扩展性不佳:DAP 可将内核工作负载减少高达 8 倍。小型工作负载主要由启动和完成任务组成,因此 GPU 带宽不饱和,这使得内核可扩展性更加糟糕。
- CPU 使用度:OpenFold 中有超过 15 万次运算。许多小型连续内核会导致 PyTorch 运算启动产生重大开销。
在图 1 中,灰色数据仅显示 OpenFold 和 DAP 增强 OpenFold (灰色虚线)。提供额外速度的 GPU 最大数量为 512,速度约为配备 8 个 GPU 的一个节点的 40 倍。
使用 NVIDIA GPU 优化 OpenFold 训练
为应对这些挑战, NVIDIA MLPerf HPC 基准测试团队进行了一系列系统优化:
- 引入了新的非阻塞数据管道,以解决慢速工作问题
- 识别关键计算模式,例如多头注意力、层数范数、随机权重平均 (SWA) 和 OpenFold 训练中的梯度裁剪。然后,该团队为每种模式设计了专用的内核,在整个 OpenFold 模型中融合了碎片计算,并针对各种工作负载大小和目标硬件架构仔细调整了内核配置。
- 添加并应用了完整的 bfloat16 支持 CUDA 图形 因为它无需与 CPU 进行交互,从而更大限度地减少了启动操作的开销。
OpenFold 的训练过程
批量大小设置为 128,以便在搭载 DAP8 的 1056 NVIDIA H100 GPU 上训练前 5K 步长,其中 1024 个 GPU 用于训练,32 个 GPU 用于评估。在每种情况下,在执行前 5K 训练步骤之前,训练指标必须超过 0.8 lDDT-C。
全局批量大小已切换到 256 ( NVIDIA Triton mha 内核已禁用),以便在搭载 DAP4 的 1056 NVIDIA H100 GPU 上训练其余步骤(1024 GPU 用于训练,32 GPU 用于评估)。初始训练过程总共需要 12 小时。
或者,您可以使用搭载 DAP8 的 2080 NVIDIA H100 GPU (2048 GPU 用于训练,32 GPU 用于评估)来训练其余步骤,初始训练时间可缩短至 10 小时。
使用 OpenFold 加速蛋白质结构
通过将其与一系列细粒度优化相结合,整体通信效率得到大幅提升。可扩展性问题得到成功解决,并将 OpenFold 训练扩展到 2080 NVIDIA H100 GPU,而之前的工作仅扩展到 512 NVIDIA A100 GPU.在 MLPerf HPC v3.0 基准测试中,NVIDIA 优化的 OpenFold 在 7.51 分钟内完成了部分训练任务 比基准测试快 6 倍以上。
在图 1 中,绿色数据显示了相同的训练,使用 DAP 和这些优化实现了 4 倍的可扩展性提升到 2048 GPU,速度提高了 4.5 倍,比运行通用 OpenFold 的一台 8 GPU 服务器的速度快 180 倍。
经过训练优化的 OpenFold 需要 5 万到 6 万步才能达到 0.9 lDDT-C,使用 1056 NVIDIA H100 GPU 需要 12.4 小时,使用 2080 NVIDIA H100 GPU 需要 10 小时。与原始 AlphaFold2 所需的 7 天相比,这是一项重大改进,与以前的工作相比,创下了新纪录。
适用于生命科学的生成式 AI 工具包
我们希望这项工作能够通过提供有效的机制来扩展基于深度学习的计算方法来解决蛋白质折叠问题,从而使 HPC 和生物信息学研究社区受益。
我们还希望这项工作中使用的工作负载分析和优化方法能够说明机器学习系统的设计和实现。这项工作中的优化可以应用到其他类似 AlphaFold2 的模型。
OpenFold 模型的版本和 NVIDIA 用于训练该模型的软件将很快在 NVIDIA BioNeMo 以及相关的 framework 用于药物研发的生成式 AI 平台。
有关更多信息,请参阅以下资源:





