本文将深入分析 DICOM 医学影像的解码功能。AWS HealthImaging 利用 NVIDIA 的 nvJPEG2000 库 来实现此功能。我们将深入探讨图像解码的复杂性,并介绍 AWS HealthImaging,以及 GPU 加速解码解决方案带来的进步。
通过 GPU 加速的 nvJPEG2000 库,踏上在 AWS HealthImaging 中提高吞吐量和降低医疗影像解密成本的旅程,代表着在云环境中实现运营效率的一大步。这些创新有望节省大量成本,预测表明此类工作负载的潜在成本降低总计数亿美元。
JPEG 2000
JPEG 2000 的实施面臨著顯著的複雜性,因為早期遇到的互操作性問題阻礙了跨不同系統的無縫整合。這種複雜性導致廣泛採用的阻礙。然而,高吞吐量 JPEG 2000 (HTJ2K) 編碼系統的出現代表著圖像壓縮技術的重大進展。JPEG 2000 標準的第 15 部分概述的 HTJ2K 利用更有效的 FBCOT (优化截断的快速块编码) 替代原始块编码算法 EBCOT (优化截断的嵌入式块编码),以提高吞吐量。
这项新标准解决了解码速度的限制,并为 JPEG 2000 在医学影像领域的更广泛采用打开了大门。HTJ2K 同时支持无损压缩和无损压缩,在保留关键医疗细节和实现高效存储之间实现了平衡。具有任意宽度和高度的灰度和彩色图像,以及每个通道多达 16 位的支持,展示了 HTJ2K 的适应性。新标准对分解级别没有限制,支持广泛的选项。
nvJPEG2000 库
随着 GPU 加速技术的进步,nvJPEG2000 进一步提高了 HTJ2K 的解码性能。这种进步释放了 JPEG 2000 在医学影像处理中的真正潜力,使其成为医疗健康提供商、研究人员和开发人员可行且高效的解决方案。nvJPEG2000 提供一个 C API 包括用于解码单个图像的 nvjpeg2kDecode 和用于解码图像中特定图块的 nvjpeg2kDecodeTile 等函数。该库提供了:
- 统一 API 接口 nvImageCodec:**该开源库与 Python 无缝集成,为开发者提供了便捷的界面。
- 解码性能分析:HTJ2K 与传统 JPEG 2000 的解码性能进行比较分析,深入了解 GPU 加速的机制。
为了确保可用性、高性能以及生产准备,本文将探讨如何将 HTJ2K 解码与 MONAI 框架结合起来。MONAI 是一种专为医学影像分析设计的框架。MONAI 部署应用 SDK 提供高性能功能,并有助于在医学影像 AI 应用程序中进行调试。本文还深入探讨了使用 AWS HealthImaging、MONAI 和 nvJPEG2000 进行医学影像处理所带来的成本效益。
采用 AWS HealthImaging 的企业级医学影像存储
得益于无损 HTJ2K 编码和 AWS 高性能网络主干,AWS HealthImaging 提供亚秒级图像检索,并可快速访问云中图像。它与工作流程无关,可无缝集成到现有的医学成像工作流程中。它符合 DICOM 标准,确保在医学影像通信中具有互操作性并符合行业标准。该服务提供本地 API,以实现可扩展和快速的图像提取,以适应不断增长的医学成像数据量。
GPU 加速的图像解码
为进一步增强图像解码性能,AWS HealthImaging 专门利用 NVIDIA nvJPEG2000 库支持 GPU 加速。此 GPU 加速可确保快速高效地对医学影像进行解码,使医疗健康提供商能够以前所未有的速度访问关键信息。HTJ2K 解码的支持功能包含广泛的选项,可适应不同的图像类型、大小、压缩需求和解码场景,使其成为各种图像处理应用程序的通用和适应性选择。这些功能包括:
- 图像格式:HTJ2K 支持任意宽度和高度的灰度和彩色图像,可适应各种图像格式和尺寸。
- 位深:HTJ2K 支持每通道高达 16 位深度的图像,确保准确呈现颜色和细节。
- 无损压缩:HTJ2K 标准支持无损压缩,确保在不丢失任何数据的情况下保持画质。
- 统一代码块配置:HTJ2K 中所有代码块都符合 HT (高吞吐量) 标准,无需进行优化代码块,以简化解码过程。
- 代码块大小:HTJ2K 利用不同的代码块大小,例如 64 × 64、32 × 32 和 16 × 16.这种可适应性支持高效地表示细节和复杂性各不相同的图像。
- 进度顺序:HTJ2K 支持多种进度顺序,包括: – LRCP (层分辨率 – 组件 – 位置) – RLCP (分辨率层组件位置) – RPCL (分辨率位置组件层) – PCRL (位置 – 组件 – 分辨率层) – CPRL (组件位置分辨率层)
- 变量分解水平:该标准允许不同的分解级别,从 1 到 5 个不等。这种分解灵活性提供了根据特定需求优化图像压缩的选项。
- 具有不同块大小的多块解码:HTJ2K 支持解码分为多个不同大小的图块图像,以增强高效解码的能力。
AWS HealthImaging 演练
在此演示中,我们展示了 AWS HealthImaging 的使用情况。我们演示了利用 GPU 加速接口使用 SageMaker 多模型端点进行图像解码的过程。
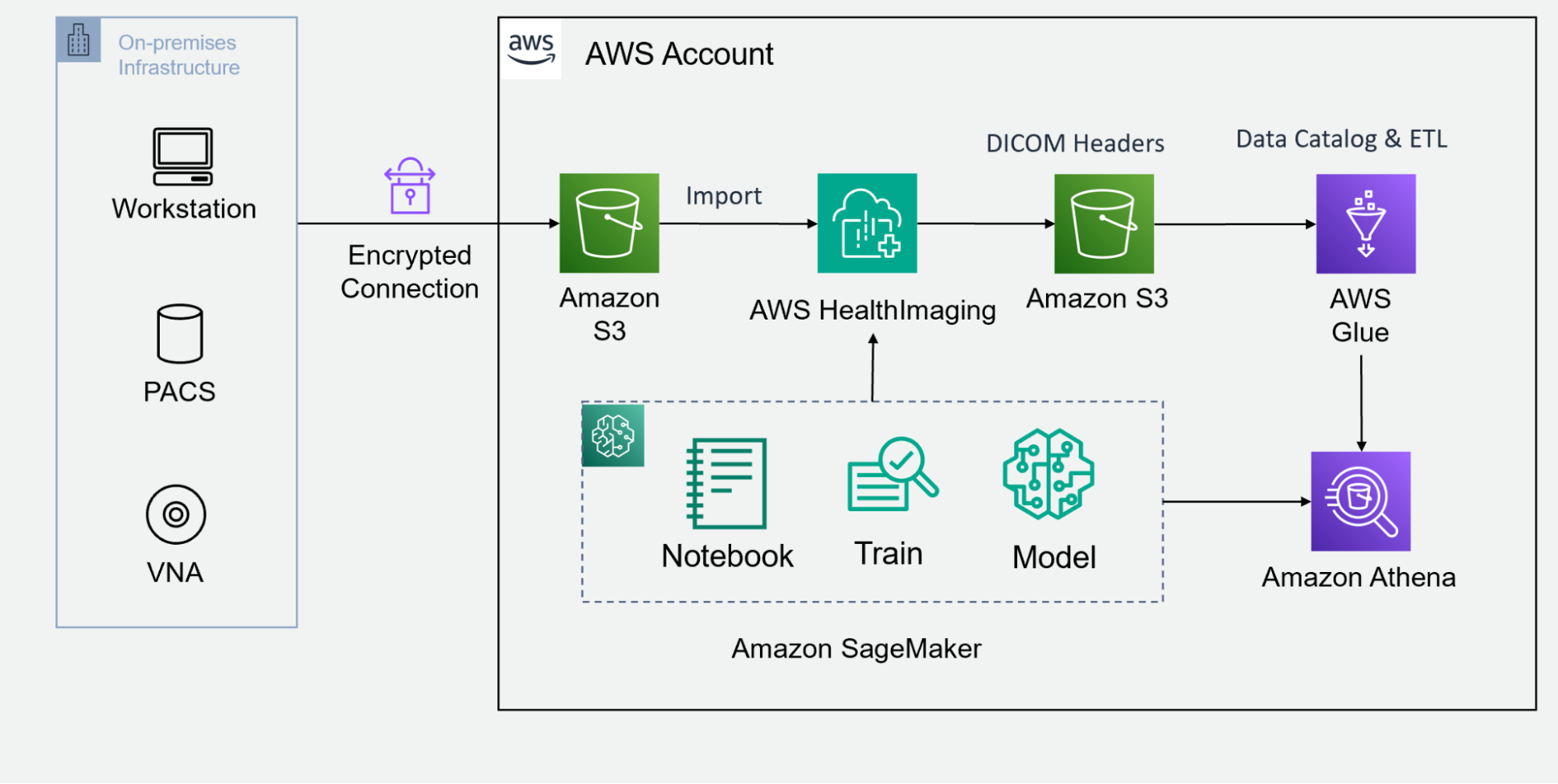
第 1 步:暂存 DICOM 图像
首先,将您的 DICOM 图像暂存到 Amazon S3 存储桶中。AWS HealthImaging 与合作伙伴产品集成,为您提供各种工具,以在指定的 S3 存储桶中上传和整理您的 DICOM 图像数据。您可以在 AWS 开放数据计划 公开的 S3 存储桶中找到包含合成医学成像数据的开放数据集,例如 合成一致性。
第 2 步:调用 API 以导入 DICOM 数据
在 S3 存储桶中暂存 DICOM 影像后,下一步是调用原生 API 将 DICOM 数据导入 AWS HealthImaging.此托管 API 有助于实现流畅的自动化流程,从而确保您的医学影像数据得到高效传输,并为进一步优化做好准备。
第 3 步:在数据湖中索引 DICOM 标头
成功导入后,从 AWS HealthImaging 中检索 DICOM 标头,解压缩数据 Blob,并将这些 JSON 对象写入 数据湖 S3 存储桶。从这里,您可以利用 AWS 数据湖分析 工具,例如: – 亚马逊胶 生成数据目录。 – 亚马逊 Athena 执行临时 SQL 查询。 – Amazon QuickSight 来构建数据可视化控制面板。您还可以将图像元数据与 其他健康数据模式 相结合,以执行多模态数据分析。
第 4 步:访问医学影像数据
借助托管 API,访问将成为无缝体验。AWS HealthImaging 可让您以亚秒级的速度以高性能和精细的方式访问成像数据。
AWS 合作伙伴的 PACS 查看器和 VNA 云端解决方案可以将图像查看应用程序与 AWS HealthImaging 集成。这些应用程序经过优化,以提供用户友好且高效的体验,以大规模查看和分析医学影像。AWS 合作伙伴 PACS 的示例包括 Allina Health、图像成像和 Visage Imaging。
科学家和研究人员可以利用 Amazon SageMaker 来执行 AI 和 ML 建模,以获得高级见解,并自动执行审查和标注任务。Amazon SageMaker 与 MONAI 可用于开发强大的 AI 模型。使用 Amazon SageMaker notebook,用户可以从 AWS HealthImaing 中检索像素帧,并使用开源工具 (例如 itkwidget) 创建 SageMaker 托管训练作业 或 模型托管端点。
作为符合 HIPAA 标准的服务,AWS HealthImaging 提供灵活性,允许远程用户安全访问和审计医疗影像数据。访问控制由 Amazon 身份和访问管理 管理,确保授权用户对 ImageSet 数据 的访问被精细控制。访问活动也可以通过 Amazon CloudTrail 跟踪。
第 5 步:支持 GPU 的 HTJ2K 解码
在典型的 AI 或 ML 工作流程 (CPU 解码路径) 中,HTJ2K 编码的像素帧将加载到 CPU 显存中,然后解码并转换为 CPU 中的张量。GPU 可以复制和处理这些像素。nvJPEG2000 可以从 AWS HealthImaging 中提取已编码的像素,并直接将其解码为 GPU 显存 (GPU 解码路径),而 MONAI 具有内置功能,可将图像数据转换为可随时输入到深度学习模型中的张量。与 CPU 解码方法相比,它的路径更短,如图 2 所示。
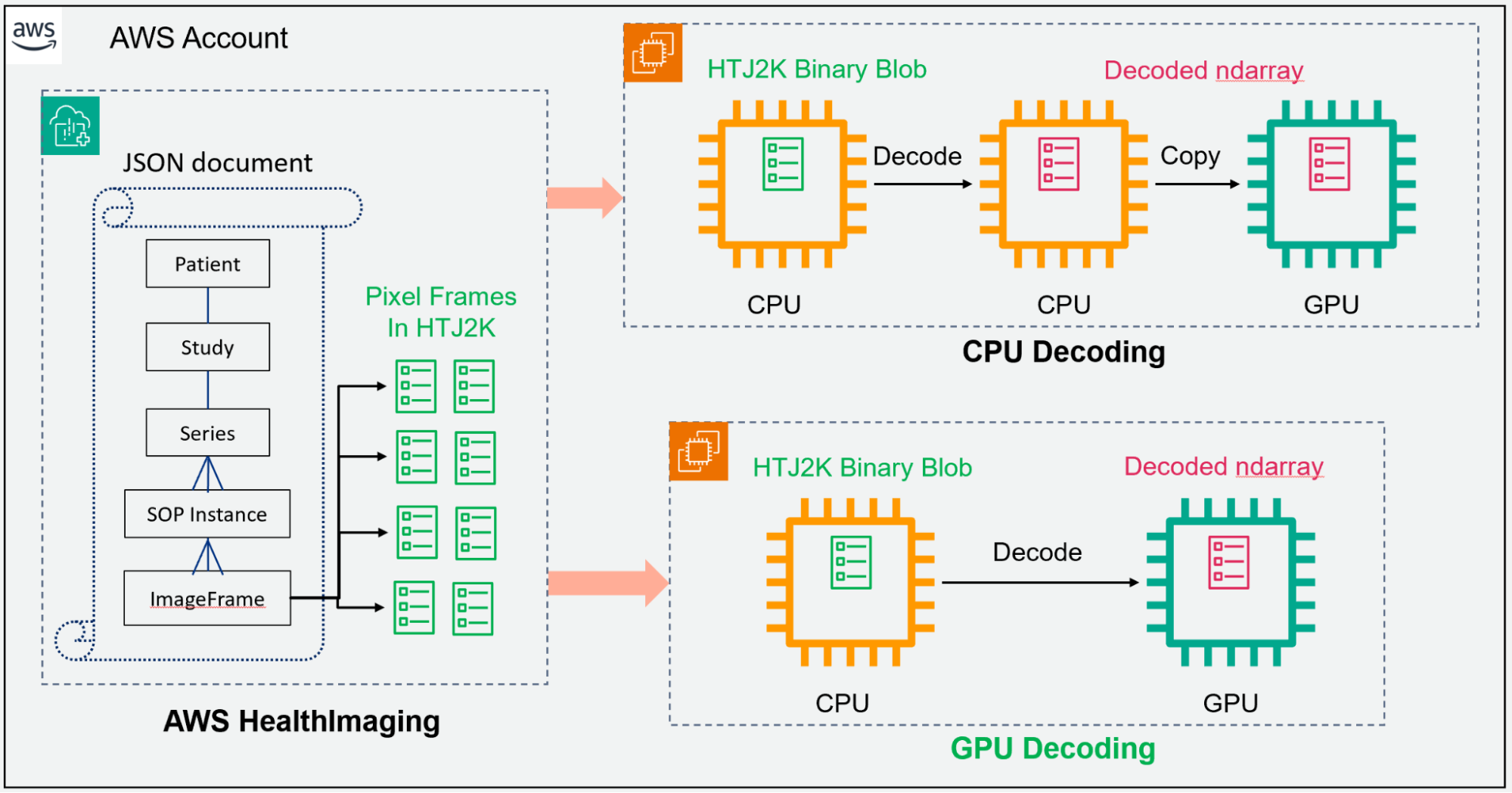
此外,nvJPEG2000 的 GPU 加速可显著提高解码性能,降低延迟并增强整体响应速度。该库与 Python 无缝集成,为开发者提供熟悉且强大的环境来执行图像解码任务。
演示 Notebook,运行在 Amazon SageMaker 上,展示了如何以可扩展且高效的方式集成和利用 GPU 加速图像解码的强大功能。在我们的实验中,SageMaker g4dn.2 xlarge 实例上的 GPU 解码速度比 SageMaker m5.2 xlarge 实例上的 CPU 解码速度快 7 倍 (图 3)。
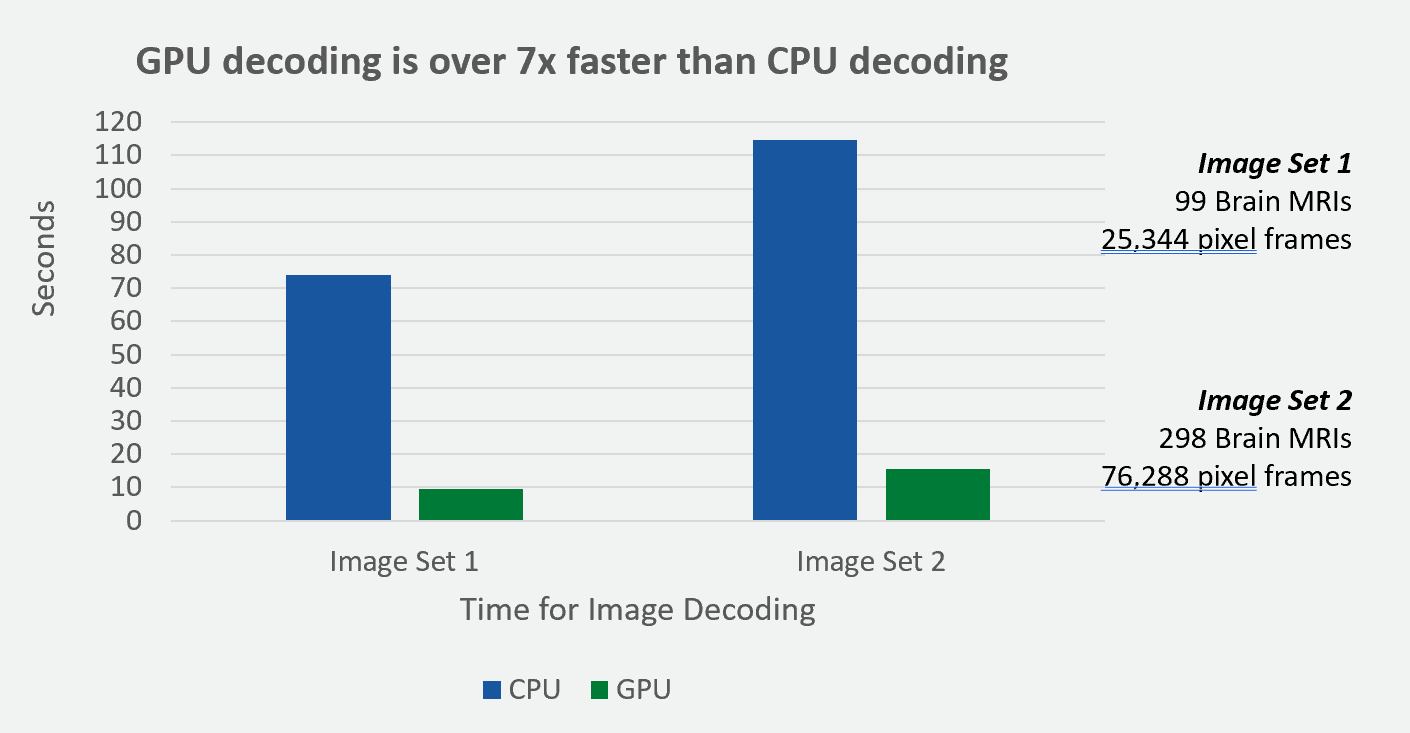
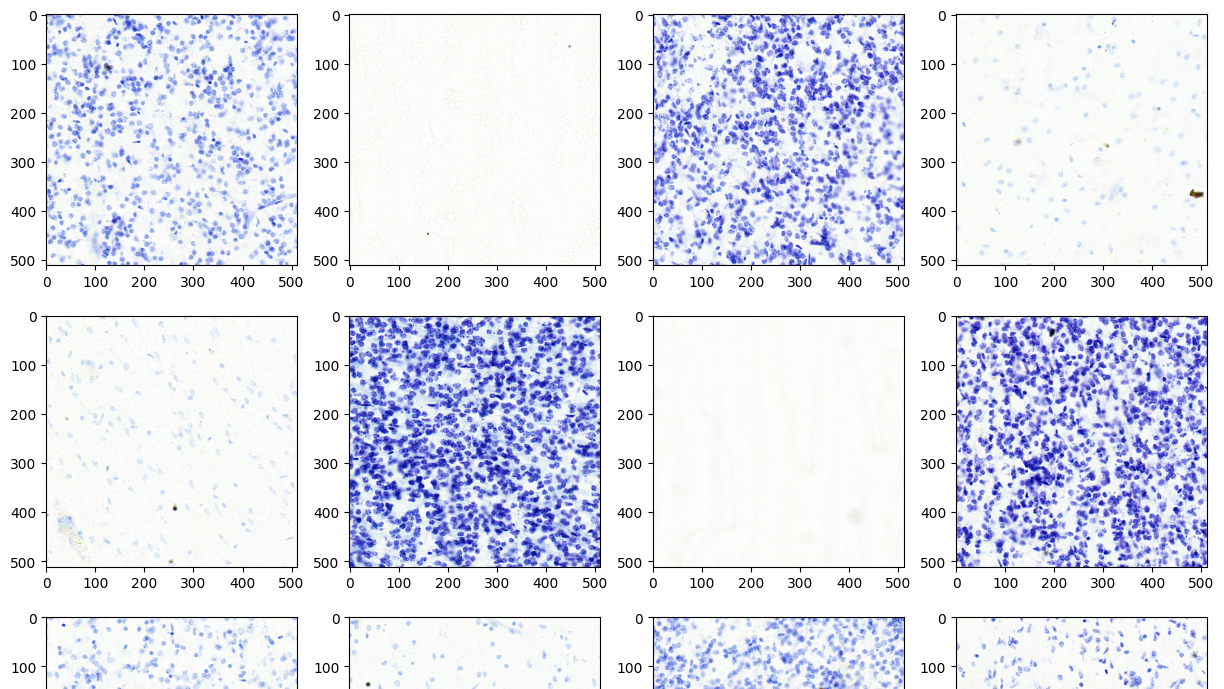
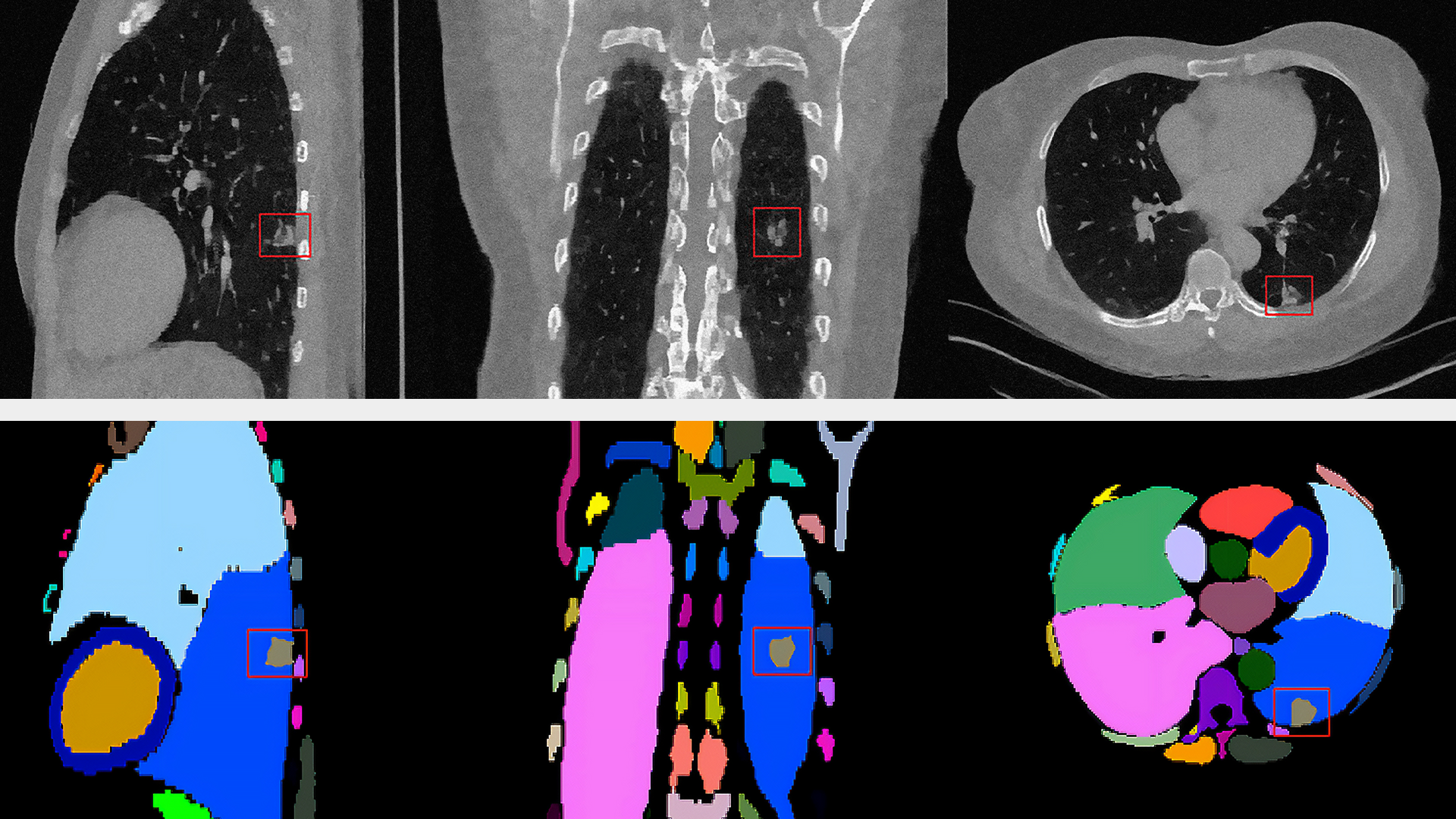
本实验中,我们使用了 合成一致性 数据集。对于不同大小的数据集,GPU 加速表现出相似的加速系数。上面标记的图像集包含脑 MRI 和像素帧。这些像素帧表示 DICOM MRI 图像,并以压缩的 HTJ2K 数据格式进行编码。
成本效益分析
AWS HealthImaging 与先进的图像解码技术相结合,不仅能提高效率,还能为医疗保健组织提供经济高效的解决方案。所提议的解决方案具有巨大的端到端成本优势,特别是考虑到通过 GPU 加速实现的惊人吞吐量加速。
单台设备的加速 NVIDIA T4 GPU 在 EC2 G4 实例上的性能提升约为 CPU 基准的 5 倍,而 EC2 G6 实例上的新 L4 GPU 通过使用多个 GPU 实例进行扩展,性能表现出近乎线性的可扩展性,在四个 NVIDIA T4 GPU 和 4个 NVIDIA L4 GPU GPU 实例上分别达到 19 倍和 48 倍。
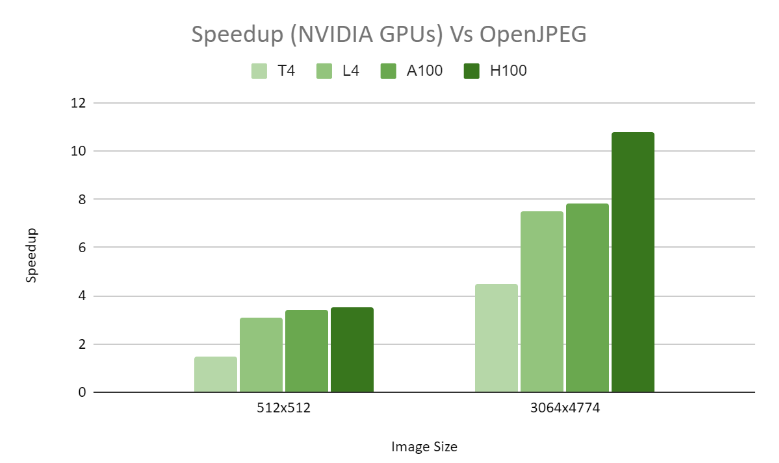
在解码性能方面,我们使用 OpenJPEG 进行了比较分析。对于 CT1 16 位 512 × 512 灰度图像,我们注意到不同 GPU 配置的速度显著提高了 2.5 倍。此外,对于尺寸为 3064 × 4774 的 MG1 16 位灰度图像,我们在各种 GPU 设置中实现了惊人的 8 倍速度提升。
为了全面评估年度云成本和能源使用情况,我们根据标准分割工作负载进行计算。此工作负载涉及每分钟向 MONAI 服务器平台上传 500 个 DICOM 文件。我们的成本估算目前仅考虑 T4 GPU,预计未来将使用 L4 GPU。我们假设使用 Amazon EC2 G4 实例。
在这种情况下,在单个 T4 GPU 上处理 DICOM 工作负载的年度成本估计约为 7400 万美元,而与 CPU 流程相关的成本为 3.454 亿美元。这意味着云支出大幅减少,预测表明此类医院工作负载可能会节省数亿美元。
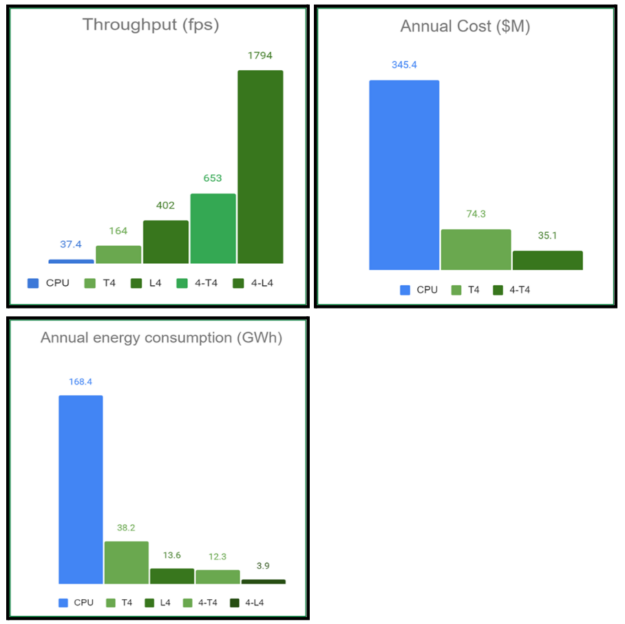
在单个 T4 GPU 上,与 CPU 基准相比,端到端吞吐量加速大约快 5 倍。在新的 L4 GPU 上,这种加速进一步提升到大约快 12 倍。当使用多个 GPU 实例时,性能几乎呈线性扩展。例如,使用 4 个 T4 GPU 时,加速大约达到 19 倍,而使用 4 个 L4 GPU 时,加速大约快 48 倍。
考虑到对环境的影响,能效是数据中心处理大型工作负载的关键因素。我们的计算表明,使用基于 GPU 的相应硬件时,以 GWh 为单位的年能耗显著降低。具体来说,单个 L4 系统的能耗约为 CPU 服务器的十二分之一。
对于类似于示例 DICOM 视频场景 (每分钟 500 小时的视频) 的工作负载,预计每年可节省数百 GWh 的能源。这些能源节省不仅具有经济效益,而且还具有重大的环境意义。温室气体排放量的减少量相当可观,类似于避免每年行驶的成千上万辆客车排放,每辆车每年约覆盖 11000 英里。
为何选择 nvImageCodec?
NVIDIA 提供 nvImageCodec 库,为开发者提供用于图像解码任务的可靠高效解决方案。nvImageCodec 利用 NVIDIA GPU 的强大功能,可提供加速解码性能,非常适合需要高吞吐量和低延迟的应用程序。
主要特性
- GPU 加速:nvImageCodec 的主要特点之一是其 GPU 加速功能。利用 NVIDIA GPU 的计算能力,nvImageCodec 可有效提高图像解码速度,从而更快地处理大型数据集。
- 无缝集成:*nvImageCodec 与 Python 无缝集成,为开发者提供熟悉的图像处理工作流环境。借助用户友好型 API,将 nvImageCodec 集成到现有 Python 项目中非常简单。
- 高效性能:借助优化的算法和并行处理,nvImageCodec 提供卓越的性能,即使在处理复杂的图像解码任务时也是如此。它支持 JPEG、JPEG 2000、TIFF 等多种图像格式,并确保快速高效的处理。
- 通用性:nvImageCodec 支持从医学成像到计算机视觉应用的各种用例。它支持处理灰度图像和彩色图像,并提供通用性和灵活性,以满足您的图像解码需求。
用例
- 医学成像:在医学影像领域,快速准确的图像解码对于及时诊断和治疗至关重要。借助 nvImageCodec,医疗保健专业人员可以快速准确地解码医学影像,从而加快决策制定并改善患者治疗效果。
- 计算机视觉:在计算机视觉应用中,图像解码速度在物体检测和图像分类等实时处理任务中至关重要。通过利用 nvImageCodec 的 GPU 加速,开发者可以实现高效的图像解码,从而提高其应用的响应速度。
- 遥感:在遥感应用中,快速高效地解码大型卫星图像对于环境监测和灾害管理等各种任务至关重要。借助 nvImageCodec,研究人员和分析人员可以轻松解码卫星图像,从而实现及时的分析和决策制定。
如何获取 nvImageCodec
获取 nvImageCodec 非常简单。您可以从以下来源获取: – PyPI – NVIDIA 开发者领地 – GitHub 下载后,您可以开始尝试编码和解码示例,以提高图像编解码器管线的效率。
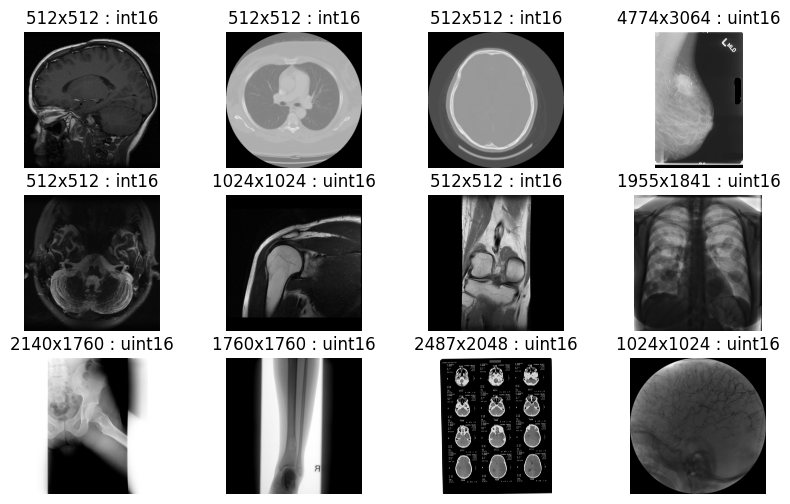
如何批量解码高吞吐量的 JPEG 2000 医学影像
以下是一个 Python 示例,展示了 批量图像解码 使用 nvImageCodec 库。此示例说明了如何使用 nvImageCodec 对 HTJ2K 图像进行批量解码。指定文件夹中的所有图像均以无损 HTJ2K 格式压缩,精度为 uint16 位。输出确认所有医学影像均已成功解码,且无损质量 (图 8)。
import os; import os.pathfrom matplotlib import pyplot as pltfrom nvidia import nvimgcodecdir = "htj2k_lossless"image_paths = [os.path.join(dir, filename) for filename in os.listdir(dir)]decode_params = nvimgcodec.DecodeParams(allow_any_depth = True, color_spec=nvimgcodec.ColorSpec.UNCHANGED)nv_imgs = nvimgcodec.Decoder().read(image_paths, decode_params)cols= 4rows = (len(nv_imgs)+cols-1)//colsfig, axes = plt.subplots(rows, cols); fig.set_figheight(2*rows); fig.set_figwidth(10)for i in range(len(nv_imgs)): axes[i//cols][i%cols].set_title("%ix%i : %s"%(nv_imgs[i].height, nv_imgs[i].width, nv_imgs[i].dtype)); axes[i//cols][i%cols].set_axis_off() axes[i//cols][i%cols].imshow(nv_imgs[i].cpu(), cmap='gray') |
如何批量解码多个 JPEG 2000 图块
以下是一个 Python 示例,展示了 基于块的图像解码 使用 nvImageCodec 库处理大型图像。这个示例展示了如何使用 nvImageCodec 解码相当大小的 JPEG 2000 压缩图像的过程。每个图块代表一个感兴趣的区域 (ROI),其大小为 512 x 512 像素。
解码过程包括将图像分割为图块,确定区域总数,然后使用 nvImageCodec 根据每个图块的索引对其进行解码,从而提供特定的图块解码信息。生成的输出会显示与不同图块相关的信息。
from matplotlib import pyplot as pltimport numpy as npimport random; random.seed(654321)from nvidia import nvimgcodecjp2_stream = nvimgcodec.CodeStream('./B_37_FB3-SL_570-ST_NISL-SE_1708_lossless.jp2')def get_region_grid(stream, roi_height, roi_width): regions = [] num_regions_y = int(np.ceil(stream.height / roi_height)) num_regions_x = int(np.ceil(stream.width / roi_width)) for tile_y in range(num_regions_y): for tile_x in range(num_regions_x): tile_start = (tile_y * roi_height, tile_x * roi_width) tile_end = (np.clip((tile_y + 1) * roi_height, 0, stream.height), np.clip((tile_x + 1) * roi_width, 0, stream.width)) regions.append(nvimgcodec.Region(start=tile_start, end=tile_end)) print(f"{len(regions)} {roi_height}x{roi_width} regions in total") return regionsregions_native_tiles = get_region_grid(jp2_stream, jp2_stream.tile_height, jp2_stream.tile_width) # 512x512 tilesdec_srcs = [nvimgcodec.DecodeSource(jp2_stream, region=regions_native_tiles[random.randint(0, len(regions_native_tiles)-1)]) for k in range(16)]imgs = nvimgcodec.Decoder().decode(dec_srcs)fig, axes = plt.subplots(4, 4)fig.set_figheight(15)fig.set_figwidth(15)i = 0for ax0 in axes: for ax1 in ax0: ax1.imshow(np.array(imgs[i].cpu())) i = i + 1 |
结束语
无论您是医疗健康提供商、研究人员还是开发者,JPEG 2000 的复兴与尖端技术一起,为医学成像的关键领域开辟了新的创新途径。AWS HealthImaging 与先进的压缩标准和 GPU 加速相结合,成为致力于增强诊断能力和提高患者治疗效果的医疗健康专业人员的重要工具。
这项创新为高性能 多模态数据分析 无缝集成基因组学、临床和医学成像数据,以提取有意义的见解。云上托管的数据科学平台简化了模型训练和部署流程,减少了摩擦。利用这些进步,并通过加速和可靠的图像解码来推动医疗健康行业的未来发展。
要开始使用,请探索以下资源: – nvImageCodec:用于图像编码和解码的库。 – MONAI on AWS 研讨会:有关使用 MONAI 在 AWS 上的指南。 – 借助 AWS Health 和机器学习服务进行多模态数据分析:有关使用 AWS Health 和机器学习服务进行多模态数据分析的指南。
- 使用 nvJPEG2000 库加速数字病理学和卫星图像的 JPEG 2000 解码。
- 借助 AWS 上的 MONAI 加速医学影像研究
- 使用 NVIDIA nvJPEG2000 在 AWS HealthImaging 中解码医学影像
- NVIDIA DGX 上细胞分辨率下的全人脑神经网络映射