NVIDIA BioNeMo 框架已发布,现可在 NGC 上下载,使研究人员能够在药物研发应用中构建和部署生成式 AI、大型语言模型 (LLM) 和基础模型。
BioNeMo 平台包括托管服务、API 端点和训练框架,可简化、加速和扩展用于药物研发的生成式 AI.BioNeMo 能够通过大规模端到端加速预训练或微调先进的模型。它可作为 NVIDIA DGX 云上的完全托管服务和 NVIDIA Base Command 平台,也可作为可下载的框架,用于部署本地基础架构和各种云平台。
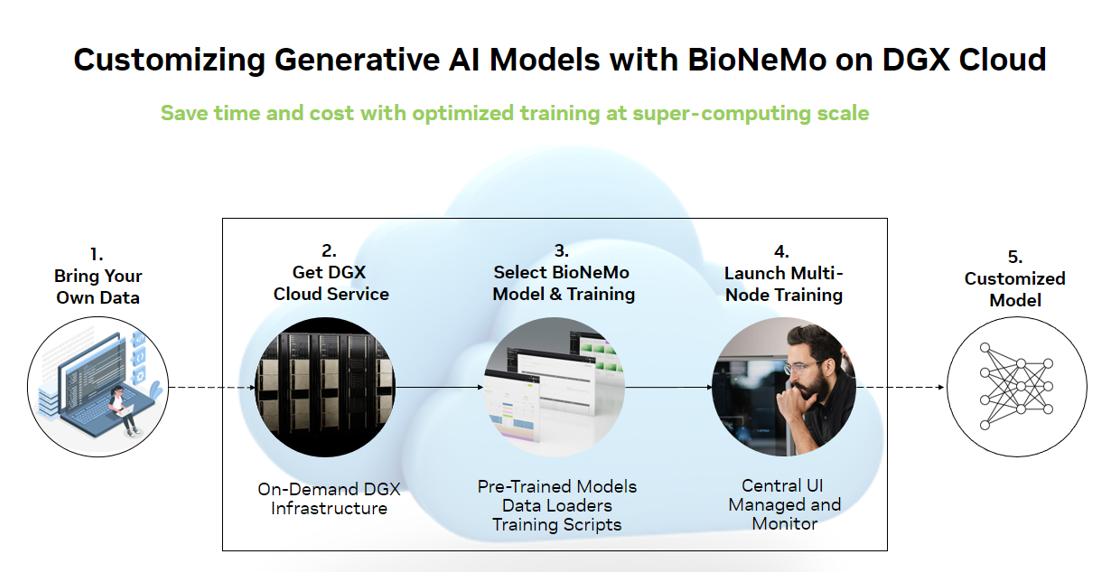
这为药物研发人员和开发者提供了一种快速轻松的方法,以便在从标识别到潜在客户优化的整个药物研发流程中构建和集成先进的 AI 应用。
BioNeMo 框架 v1.0 功能
- 轻松加载数据:具备自动下载器、预处理数据功能,以及对常见生物分子数据格式的支持。
- 特定于 SOTA 领域的模型:包括即开即用的架构和经过验证的检查点,适用于蛋白质和小分子数据的训练。
- 优化扩展方法用于在数千个 GPU 上进行无缝加速训练,并经过优化以最大化吞吐量并降低成本。
- 灵活的训练工作流程:轻松地从零开始进行大规模预训练、从可靠的检查点进行微调,以及迅速开展下游任务的训练。
- 验证在环:定期监督任务训练,用于测量模型训练过程中嵌入的质量。实现完全自动化,并与 Weights and Biases 集成。
优化蛋白质和小分子模型的训练
NVIDIA BioNeMo 为多个领域的生成式 AI 模型提供优化。BioNeMo 框架 v1.0 提供优化的模型架构和工具,用于训练蛋白质和小分子 LLM:
- BioNeMo ESM1 和 ESM2
- BioNeMo MegaMolBART
- BioNeMo ProtT5
BioNeMo ESM1 和 ESM2
ESM 模型系列是一系列基于 Transformer 的蛋白质语言模型,这些模型是基于 BERT 架构构建的,并由 Meta 基础 AI 研究蛋白质团队 (FAIR) 开发。
通用型类似 ESM 的架构已经过优化,现已在 BioNeMo 框架中提供,并可用于蛋白质 LLM 的自定义训练。这些模型基于蛋白质序列的海量数据集进行训练,以了解控制蛋白质结构和功能的氨基酸之间的基础模式和关系。
重要的是,可以通过迁移学习将经过训练的 ESM 模型用于各种下游任务。例如,您可以使用其编码器中的嵌入来训练具有监督式学习目标的较小模型,以推理蛋白质的属性。事实证明,这可以为 3D 结构预测、变异效应预测或设计等各种任务生成高度准确的模型从头开始蛋白质。
BioNeMo 框架包含针对 ESM-2 6.5 亿和 3B 参数模型的经过验证的训练检查点,可实现零启动,以创建特定领域的自定义应用程序。此外,还提供了许多下游任务示例,包括二级结构预测、亚蜂窝定位预测和热稳定性预测。
BioNeMo MegaMolBART
MegaMolBART 模型是使用 seq2seq Transformer BART 架构构建的生成化学模型,其灵感来自 AstraZeneca 开发的 Chemformer 模型。MegaMolBART 在小分子 SMILES 字符串的 ZINC-15 数据库上进行训练,总共使用 15 亿个分子进行训练。
MegaMolBART 编码器的嵌入可用于下游预测模型,这与 ESM 或编码器和解码器相同,可通过对嵌入空间进行采样来生成新分子。这意味着 MegaMolBART 可用于各种化学信息学药物研发任务,例如反应预测、分子优化和从头开始分子生成。
MegaMolBART 使用 BioNeMo 框架开发,该框架包括经过训练和验证的 4500 万参数模型检查点。下游任务工作流还用于预测回合成反应和物理化学属性,例如亲脂性、水解性 (ESOL) 和无水化能 (FreeSolv).
BioNeMo ProtT5
ProtT5 是一种基于编码器/解码器 LLM 构建的蛋白质语言模型,由 Rost Lab 使用 T5 架构开发。与 ESM 模型一样,ProtT5 可以从其编码器生成用于表征学习的嵌入,但也可以使用整个编码器/解码器架构执行序列翻译任务。
与其他模型一样,基础模型可以扩展应用到下游任务,例如生成蛋白质序列。最近的一个例子是初创公司 Evozyne 创造了两种蛋白质,这些蛋白质在医疗保健(旨在治疗先天性疾病)和清洁能源(旨在消耗二氧化碳以减少全球暖化)方面具有巨大潜力。
ProtT5 模型作为 BioNeMo 框架的一部分进行了优化,包括用于 192M 参数模型的经过训练和验证的检查点,以及用于二级结构预测的下游任务工作流程示例。
借助 BioNeMo 框架实现速度和规模
BioNeMo 框架使用各种技术来实现更高的吞吐量和可扩展性,包括并行性:
- 模型管道并行:模型层的分布式布局用于实现并行训练。
- 模型张量并行:层本身被切片并分布处理。
指定精度等优化也可以带来巨大的性能优势,通常对模型准确性几乎没有影响。
BioNeMo 框架包含用于选择和调整模型超参数的最佳实践,能够轻松配置其中许多选项以实现最大性能。其中一个示例是将模型张量并行化等技术应用于大小超过 10 亿个参数的模型,以及用于超过 50 亿个参数的模型的模型管道并行化。
借助 BioNeMo 框架跨 H100 GPU 扩展 ESM2 训练
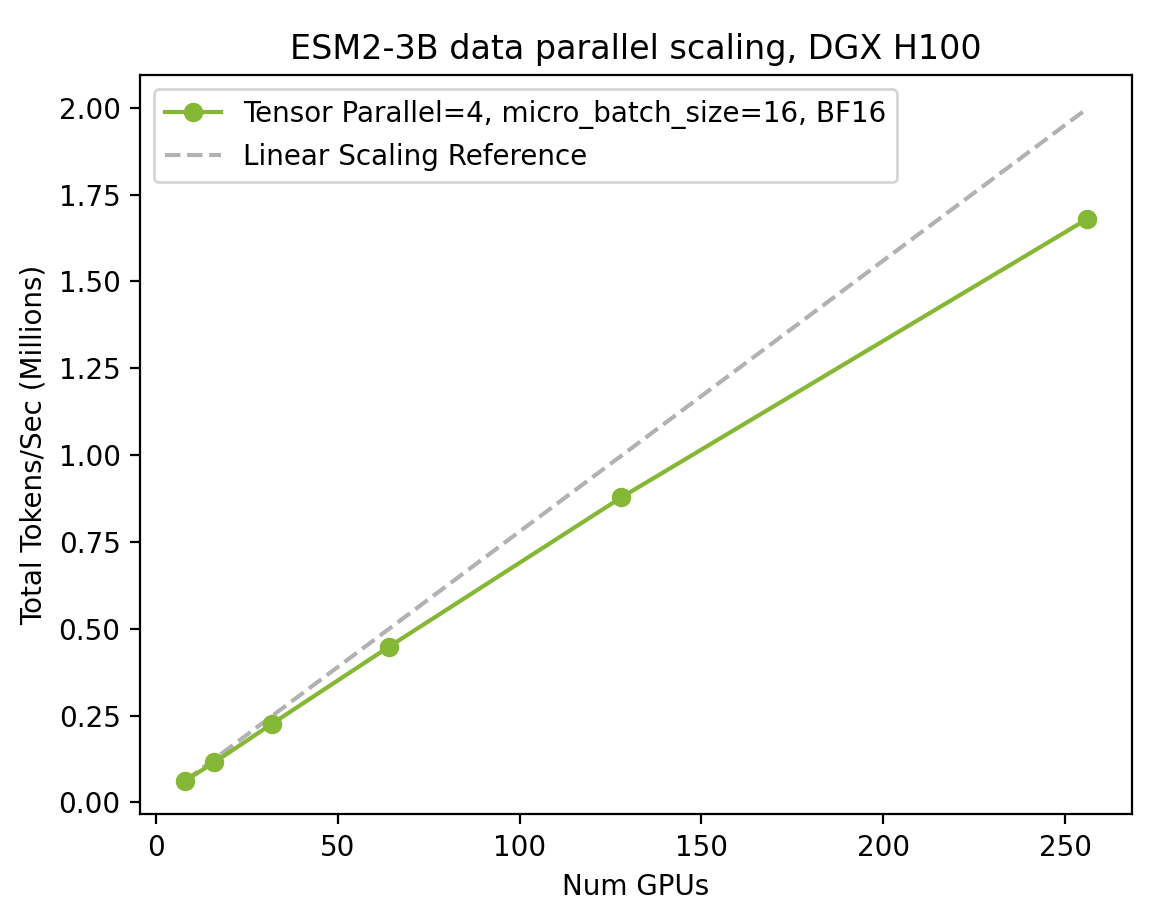
图 2 显示了从单个 DGX 节点(8 个 H100 GPU)扩展到 32 个 DGX 节点(256 个 H100 GPU),以及由此产生的吞吐量增加(每秒令牌数)。
BioNeMo 框架和最新的 NVIDIA GPU 提供的全栈优化能够以更快、更高效的速度训练先进的模型。
例如,ESM2 在 512 V100 GPU 上作为原始出版物的一部分进行训练,在 8 天内完成了 6.5 亿参数模型的训练,在 30 天内完成了 3B 参数模型的训练。现在,使用 BioNeMo 框架和 512 H100 GPU (使用 1T 令牌或 11.9 B 蛋白质序列进行训练)训练相同的模型,分别只需 1.2 天和 3.5 天。
这为在更短的时间内训练更大的模型提供了机会。例如,使用 BioNeMo 框架和 512 H100 GPU,可以在 18.6 天内使用 1T 令牌训练包含 200 亿个参数的 ESM2 模型。
在更短的时间内训练更大的 ESM2 模型
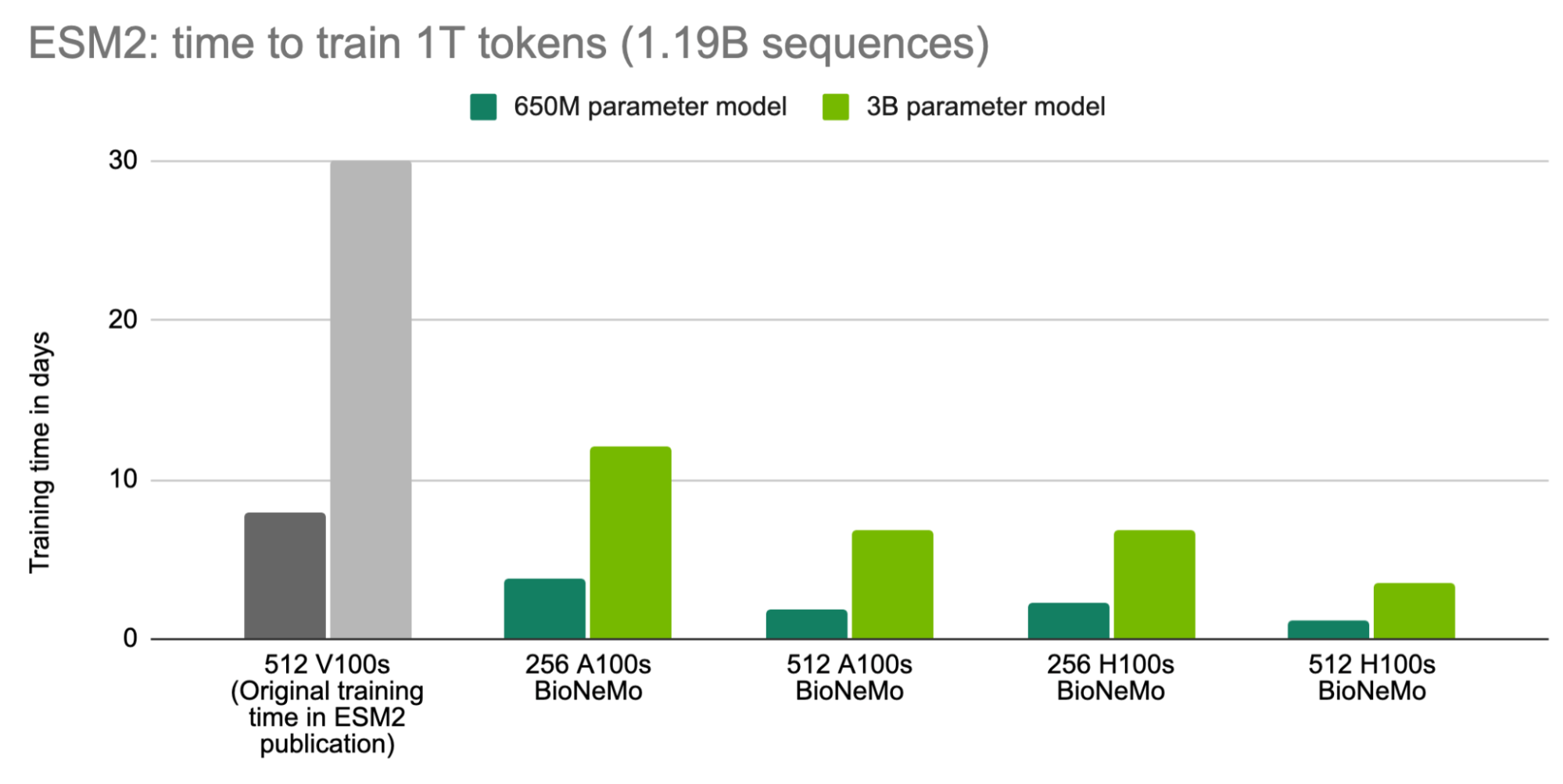
我们原始发布的模型的训练时间以灰色显示,供参考。使用 BioNeMo 训练的模型是以 1T 令牌(相当于 11.9 亿个蛋白质序列)进行训练的。
BioNeMo 框架入门
BioNeMo 框架 v1.0 现已在 NGC 上推出。欲了解如何访问、获取最新技术文章以及观看有关 AI 药物研发的演讲,请访问 BioNeMo 的开始使用页面和资源页面。
BioNeMo 框架最佳部署在 NVIDIA DGX 云上,后者可按需提供 DGX 基础设施,以实现卓越的吞吐量性能。这为云端企业级 AI 计算提供了全面的 AI 训练即服务解决方案,并可直接联系 NVIDIA AI 专家。有关更多信息,请参阅 DGX 云 页面。