在科学、技术和医学的交叉领域,寻找可行的药物是最艰巨的挑战之一。从数学上讲,随机偶然发现一个好的治疗候选者的可能性小得惊人。这主要是因为只有几个原子可以通过大量的方法连接在一起,使初看起来像药物一样的化合物。
在对这些分子进行更深入的检查后,绝大多数分子都会成为不适合的治疗方法。临床上可行的药物必须具有多种特性或特性,如果缺少其中任何一种特性或特性或超出范围,则可能会使药物无效,甚至有毒。药物猎人寻求的特性示例包括描述药物的结合亲和力、溶剂性、膜渗透性、分子量和稳定性的特性,等等。
从本质上讲,候选药物的追求是多目标优化问题。
生成式 AI 模型,例如 NVIDIA BioNeMo 中的 MolMIM,旨在直接解决寻找具有正确属性的分子的挑战。使用 MolMIM,研究人员可以生成分子,以最大限度地提高用户指定的评分函数(简称 Oracle 函数)。MolMIM 执行受控生成,在用户提供的 Oracle 函数的指导下,浏览化学空间的学习内部表示。
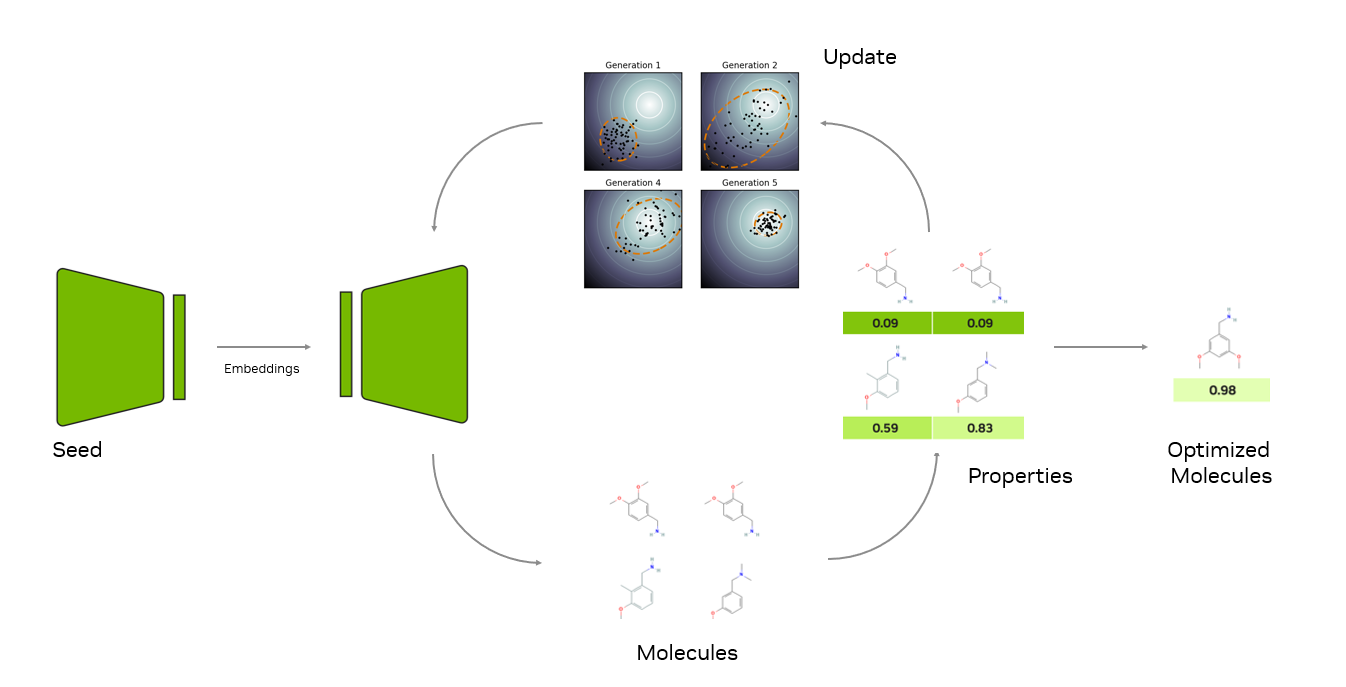
研究人员可以定义他们想要的任何目标函数,甚至是计算多个目标的函数。在幕后,MolMIM 使用名为 CMA-ES 的无梯度数值优化算法来浏览模型的潜在空间。生成过程是迭代的:
- 在每次迭代中,MolMIM 都会生成一批分子。
- 借助用户提供的 Oracle 函数,计算分子的属性。
- 然后,使用协方差矩阵适应进化策略 (CMA-ES) 更新隐向量,以生成下一批分子。
- 算法会一直持续到收。
使用 BioNeMo Cloud API 和 BioNeMo 的 Python 客户端库,优化分子生成工作流程非常简单。要定义 Oracle 函数,您只需编写一个遵循简单调用签名的 Python 函数即可。该函数应获取 SMILES 列表,并返回相同长度的分数 NumPy 数组。通过定义 Oracle 函数,可以从 BioNeMo Python 客户端实例化优化器,并开始分子生成!
您可以通过 BioNeMo 的 Cloud API 抢先体验 MolMIM,该 API 将于 2024 年 1 月 19 日发布。
此外,BioNeMo 的 Cloud API 将加速更新 DiffDock,这是一种 AI 模型,用于预测蛋白质 – 配体复合体的三维结构,这在药物研发过程中是一个关键步骤。通过 BioNeMo 的 DiffDock 更新版,研究人员能够以超过相同硬件上基准实现的 2.5 倍速度预测蛋白质 – 配体复合体的三维姿态。有关更多信息,请参阅借助 NVIDIA BioNeMo 服务为药物研发构建生成式 AI 流程。
注册抢先体验 NVIDIA BioNeMo Cloud API,或立即开始使用BioNeMo 框架进行模型训练。