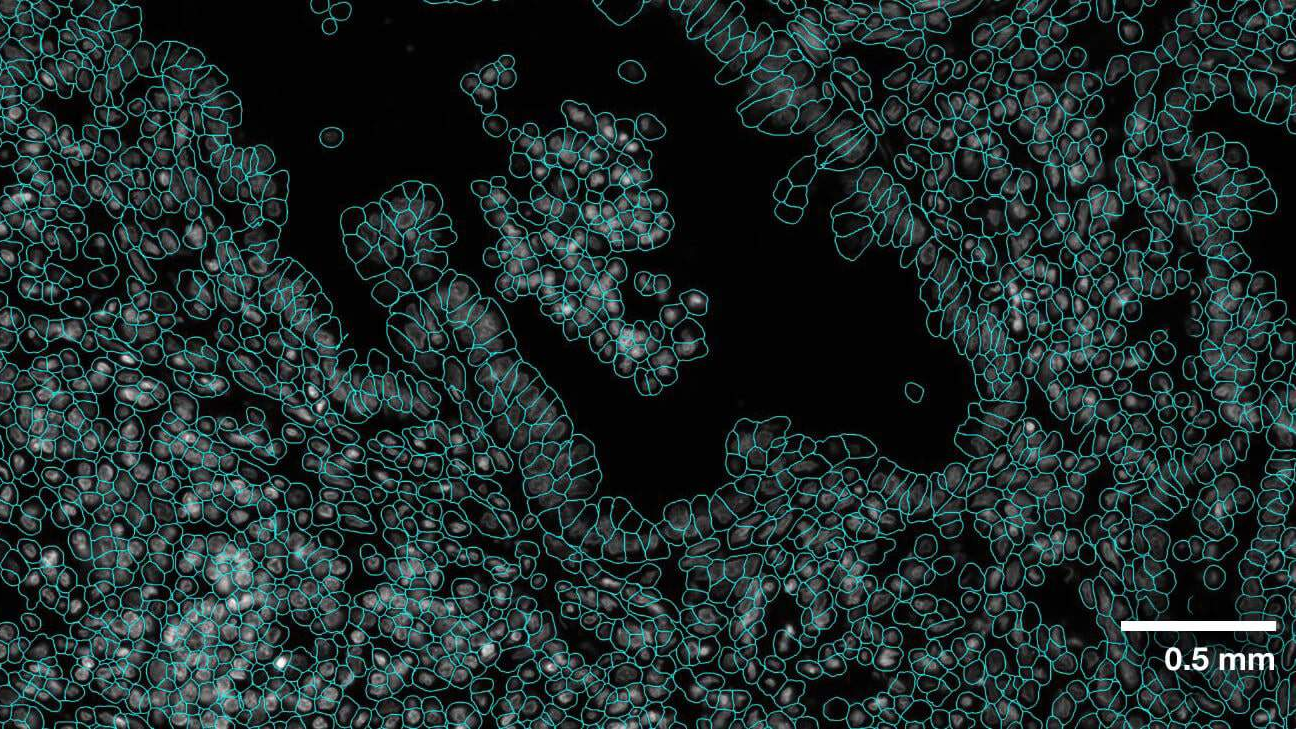
不同领域的数据科学家使用聚类方法在他们的数据集中找到自然的“相似”观察组。流行的聚类方法可以是:
- 基于质心的:根据与某个质心的接近程度将点分组为 k 组。
- 基于图形的:根据图中顶点的连接对其进行分组。
- Density-based:根据附近区域数据的密度或稀疏性更灵活地分组。
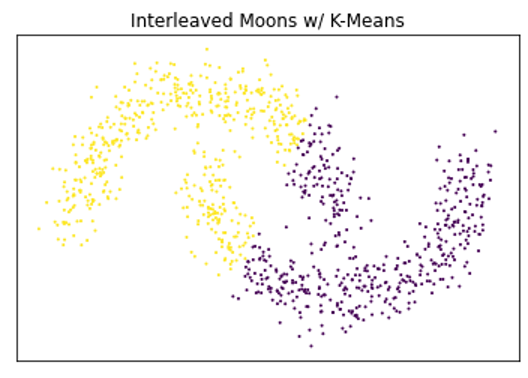
基于层次密度的应用程序空间聚类 w / Noise (HDBSCAN)算法是一种density-based聚类方法,对噪声具有鲁棒性(将稀疏区域中的点作为簇边界,并将其中一些点直接标记为噪声)。基于密度的聚类方法,如 HDBSCAN ,能够发现形状奇特、大小各异的聚类 — 与k-means、k-medioids或高斯混合模型等基于质心的聚类方法截然不同,这些方法找到一组 k 个质心,将簇建模为固定形状和大小的球。除了必须预先指定 k 之外,基于质心的算法的性能和简单性帮助它们仍然是高维聚类点的最流行方法之一;即使在不修改输入数据点的情况下,它们也无法对不同大小、形状或密度的簇进行建模。
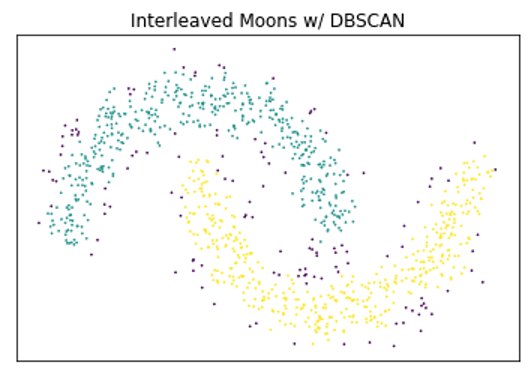
HDBSCAN 建立在一种众所周知的基于密度的聚类算法 DBSCAN 的基础上,该算法不要求提前知道簇的数量,但仍然存在一个不幸的缺点,即假设簇可以用一个全局密度阈值建模。这使得对具有不同密度的簇进行建模变得困难。 HDBSCAN 改进了这一缺点,通过使用单链聚簇来构建树状图,从而可以找到不同密度的簇。另一种著名的基于密度的聚类方法称为光学算法,它改进了 DBSCAN ,并使用分层聚类来发现密度不同的聚类。光学技术通过将点投影到一个新的空间(称为可达空间)来改进标准的单链接聚类,该空间将噪声从密集区域进一步移开,使其更易于处理。然而,与许多其他层次聚集聚类方法(如单链接聚类和完全链接聚类)一样, OPTICS 也存在以单个全局切割值切割生成的树状图的缺点。 HDBSCAN 本质上是光学+ DBSCAN ,引入了集群稳定性的度量,以在不同级别上切割树状图。
我们将通过快速示例演示 HDBSCAN 的 RAPIDS cuML 实现中当前支持的功能,并将提供我们在 GPU 上实现的一些实际示例和基准。在阅读了这篇博文之后,我们希望您对 RAPIDS ‘ GPU – 加速 HDBSCAN 实施可以为您的工作流和探索性数据分析过程带来的好处感到兴奋。
RAPIDS 中的 HDBSCAN 入门
GPU 提供了一组 RAPIDS – 加速 CPU 库,几乎可以替代 PyData 生态系统中许多流行的库。下面的示例笔记本演示了 Python 上使用最广泛的 HDBSCAN Python 库与 GPU 上的 RAPIDS cuML HDBSCAN 之间的 API 兼容性(扰流板警报–在许多情况下,它与更改导入一样简单)。
下面是一个非常简单的示例,演示了基于密度的聚类优于基于质心的技术对某些类型数据的好处,以及使用 HDBSCAN 优于 DBSCAN 的好处。
HDBSCAN 在实践中的应用
基于密度的聚类技术自然适合于许多不同的聚类任务,因为它们能够找到形状奇特、大小各异的聚类。与许多其他通用机器学习算法一样,没有免费的午餐,因此尽管 HDBSCAN 改进了一些成熟的算法,但它仍然不是完成这项工作的最佳工具。尽管如此, DBSCAN 和 HDBSCAN 在从地理空间和协同过滤/推荐系统到金融和科学计算等领域的应用中取得了显著的成功,被应用于从天文学到加速器物理学到基因组学等学科。它对噪声的鲁棒性也使得它对于异常值和异常检测应用非常有用。
与数据分析和机器学习生态系统中的许多其他工具一样,计算时间对生产系统和迭代工作流有很大的影响。更快的 HDBSCAN 意味着能够尝试更多的想法并制作更好的模型。下面是几个使用 HDBSCAN 对单词嵌入和单细胞 RNA 基因表达进行聚类的示例笔记本。这些都是为了简短,并为您自己的数据集使用 HDBSCAN 提供了一个很好的起点。您是否已成功地将 HDBSCAN 应用于工业或科学领域,我们在此未列出?请留下评论,因为我们很想听到。如果您在自己的硬件上运行示例笔记本电脑,还请告知我们您的设置以及您使用 RAPIDS 的经验。
单词嵌入
向量嵌入代表了一种流行且非常广泛的聚类机器学习应用。我们之所以选择 GoogleNews 数据集,是因为它足够大,可以很好地显示我们的算法的规模,但又足够小,可以在一台机器上执行。下面的笔记本演示了如何使用 HDBSCAN 查找有意义的主题,这些主题来自单词嵌入的角度空间中的高密度区域,并使用 UMAP 可视化生成的主题簇。它使用整个数据集的一个子集进行可视化,但为调整不同的超参数和熟悉它们对结果集群的影响提供了一个很好的演示。我们使用默认的超参数设置(形状为 3Mx300 )对整个数据集进行了基准测试,并在 24 小时后停止了 CPU 上的 Scikit learn contrib 实现。 RAPIDS 的实现大约需要 22 . 8 分钟。
单细胞 RNA
下面是一个基于扫描和俱乐部库中教程笔记本的工作流示例。本示例笔记本取自 RAPIDS 单单元示例存储库,其中还包含几个笔记本,演示了 RAPIDS 用于单细胞和三级分析。在 DGX-1 (英特尔 40 核至强 CPU + NVIDIA V100 GPU )上,我们发现 HDBSCAN (在 GPU 上是~ 1s ,而不是具有多个 CPU 线程的~ 29s )使用了包含~ 70k 肺细胞基因表达的数据集上的前 50 个主成分,加速了 29x 。
在 GPU 上加速 HDBSCAN

RAPIDS CUML 项目包括端到端 GPU 加速的 HDBSCAN ,并提供 Python 和 C ++ + API 。与 cuML 中的许多基于邻域的算法一样,它利用 Facebook 的费斯库中的蛮力 kNN 来加速相互可达空间中 kNN 图的构建。这是目前的一个主要瓶颈,我们正在研究通过精确和近似近邻选项进一步改进它的方法。
CUML 还包括单连锁层次聚类的实现,它提供了 C ++和 Python API 。 GPU – 加速单个链接算法需要计算最小生成树的新原语。此原语基于图形,因此可以在 cugraph 和 cuml 库中重用。我们的实现允许重新启动,这样我们就可以连接一个断开连接的 knn 图,并通过不必在 GPU 内存中存储整个成对距离矩阵来提高可伸缩性。
与大多数CUML算法中的C++一样,这些依赖于我们的大多数基于ML和基于图元的[VZX27 ]。最后,他们利用利兰·麦克因内斯和约翰·希利所做的伟大工作到 GPU ——甚至加快了群集压缩和选择步骤,使数据尽可能多地保留在 GPU 上,并在数据规模扩展到数百万时提供额外的性能提升。
基准
我们使用了 McInnes 等人在 CPU 上的参考实现提供的基准笔记本,将其与 cuML 的新 GPU 实现进行比较。参考实现针对低维情况进行了高度优化,我们将高维情况与大量使用 Facebook FAISS 库的暴力实现进行了比较。
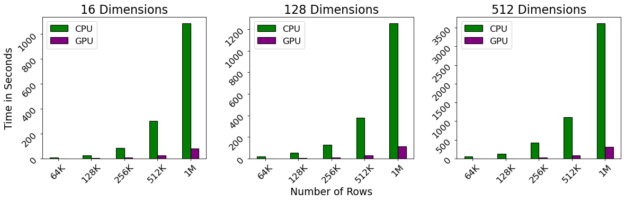
基准测试是在 DGX-1 上执行的,该 DGX-1 包含 40 核 Intel 至强 CPU 和 NVIDIA 32gb V100 GPU s 。即使对维度数进行线性缩放,对行数进行二次缩放,我们观察到 GPU 仍然保持接近交互性能,即使行数超过 1M 。

发生了什么变化?

虽然我们已经成功地在 GPU 上实现了 HDBSCAN 算法的核心,但仍有机会进一步提高其性能,例如通过加快蛮力 kNN 图构造删除距离计算,甚至使用近似 kNN。虽然欧几里德距离涵盖了最广泛的用途,但我们还想公开Scikit 学习 Contrib 实现中提供的其他距离度量。
scikit learn contrib 实现还包含许多不错的附加功能,这些功能没有包含在 HDBSCAN 上的开创性论文中,例如半监督和模糊聚类。我们也有坚固的单连杆和光学算法的构建块,这将是 RAPIDS 未来的良好补充。最后,我们希望在将来支持稀疏输入。
如果您发现这些功能中的一个或多个可以使您的应用程序或数据分析项目更成功,即使此处未列出这些功能,请转到我们的Github 项目并创建一个问题。
概括
HDBSCAN 是一种相对较新的基于密度的聚类算法“站在巨人的肩膀上”,改进了著名的 DBSCAN 和光学算法。事实上,它的核心原语还增加了重用,并为其他算法提供了构建块,例如基于图的最小生成树和 RAPIDS ML 和图库中的单链接聚类。
与其他数据建模算法一样, HDBSCAN 并不是所有工作的完美工具,但它在工业和科学计算应用中都有很多实际用途。它还可以与 PCA 或 UMAP 等降维算法配合使用,尤其是在探索性数据分析应用中。