药物研发的核心是寻求新的、有效的治疗方法,以治疗仍然对当前疗法有强烈抵抗力的疾病。随着加速计算的兴起,深度学习等 AI 技术从根本上改善了这一传统上漫长而昂贵的过程。
伦敦的药物研发公司 Receptor.AI,作为NVIDIA 初创加速计划会员,成功地将NVIDIA BioNeMo集成到其端到端计算机辅助药物研发 (CADD) 平台的云 API 中。通过这一集成,该公司实现了从基于 CPU 的传统处理到 NVIDIA 平台加速计算的转变,显著提升了包括虚拟筛选、ADMET预测和配体姿态预测在内的主要药物研发任务的效率。
本文将探讨 Receptor.ai 如何将其药物研发平台与 NVIDIA BioNeMo 相结合,以及加速计算在 ADMET 预测任务中的优势。
NVIDIA BioNeMo 作为平台的推动者
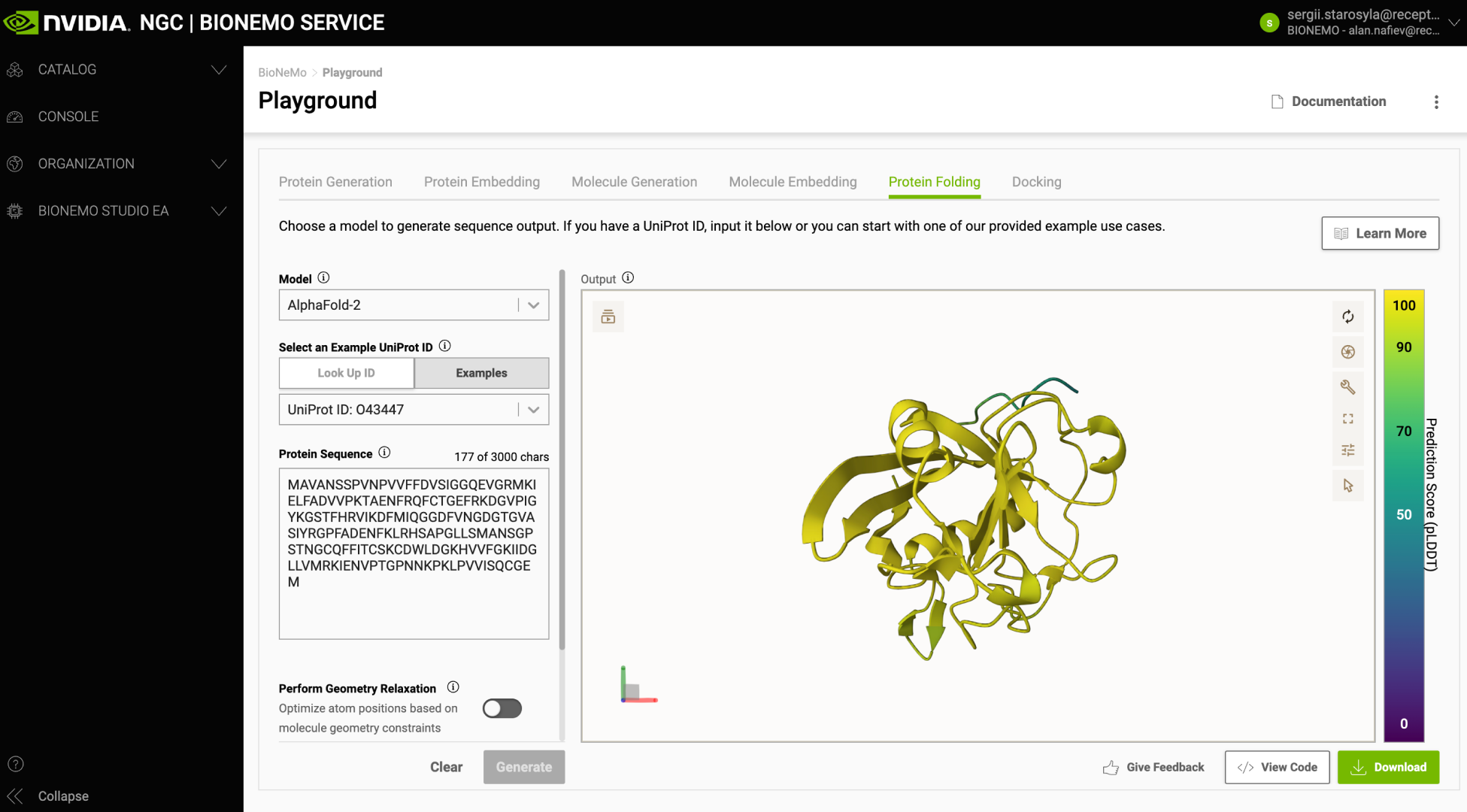
NVIDIA BioNeMo 云 API 率先提供了对药物研发至关重要的高级 AI 模型。研究人员、工程师和药物猎人可以使用这些模型来扩展和定制大型生物分子的开发和部署,这些模型包括用于蛋白质折叠、分子对接和分子生成的基础模型。BioNeMo 还包含一个 Playground(图 1),这是一个实验性的界面和参考,供开发者创建应用。
Receptor.AI 首席技术官 Sergii Starosyla 博士说:“ NVIDIA 的 BioNeMo 提供了一个基础层,其中包含高性能工具和先进的模型,可以轻松进行定制并集成到第三方药物研发工作流程中。
Starosyla 博士补充道:“通过将 BioNeMo 的强大功能与 Receptor.AI 的药物研发云平台相结合,我们可以获得由完整的 NVIDIA 硬件和软件加速堆栈支持的最新深度学习技术。这为处理极具挑战性的药物标开辟了新的机会。”
Receptor.AI 利用内部混合智能药物研发平台的强大功能,专注于设计注重选择性和向蛋白质 – 蛋白质相互作用 (PPI) 的高效疗法。
基于扩散的生物学和化学模型
Receptor.AI 利用基于扩散的生成式 AI 对药物标相互作用 (DTI) 或人体中的蛋白质与药物相互作用的方式进行建模。通过此系统,他们可以探索数十亿个化学空间,同时考虑到上和显式离蛋白的构象灵活性,以确保优化识别潜在的治疗化合物。
例如,Receptor.AI 相互作用模型可以预测分子是否与特定蛋白质相互作用,从而表明生物活动。同样,选择性预测 AI 模型评估潜在的脱相互作用,并根据数千种人类蛋白质对其选择性进行排名。Receptor.AI 的平台还评估化合物的吸收、分布、新陈代谢、排便和剧毒 (ADME-Tox),并预测多达 40 个参数,以确保安全性和有效性。
考虑到训练和服务基础模型所涉及的计算复杂性,加速计算平台已成为运行这些模型的重要组成部分。其中一个模型是进化规模建模(ESM)蛋白质序列编码模型,可通过 BioNeMo 获取,该模型为 Receptor.ai 的专有 DTI 和选择性应用提供输入功能。有关更多信息,请参阅进化规模的蛋白质序列语言模型支持准确的结构预测。
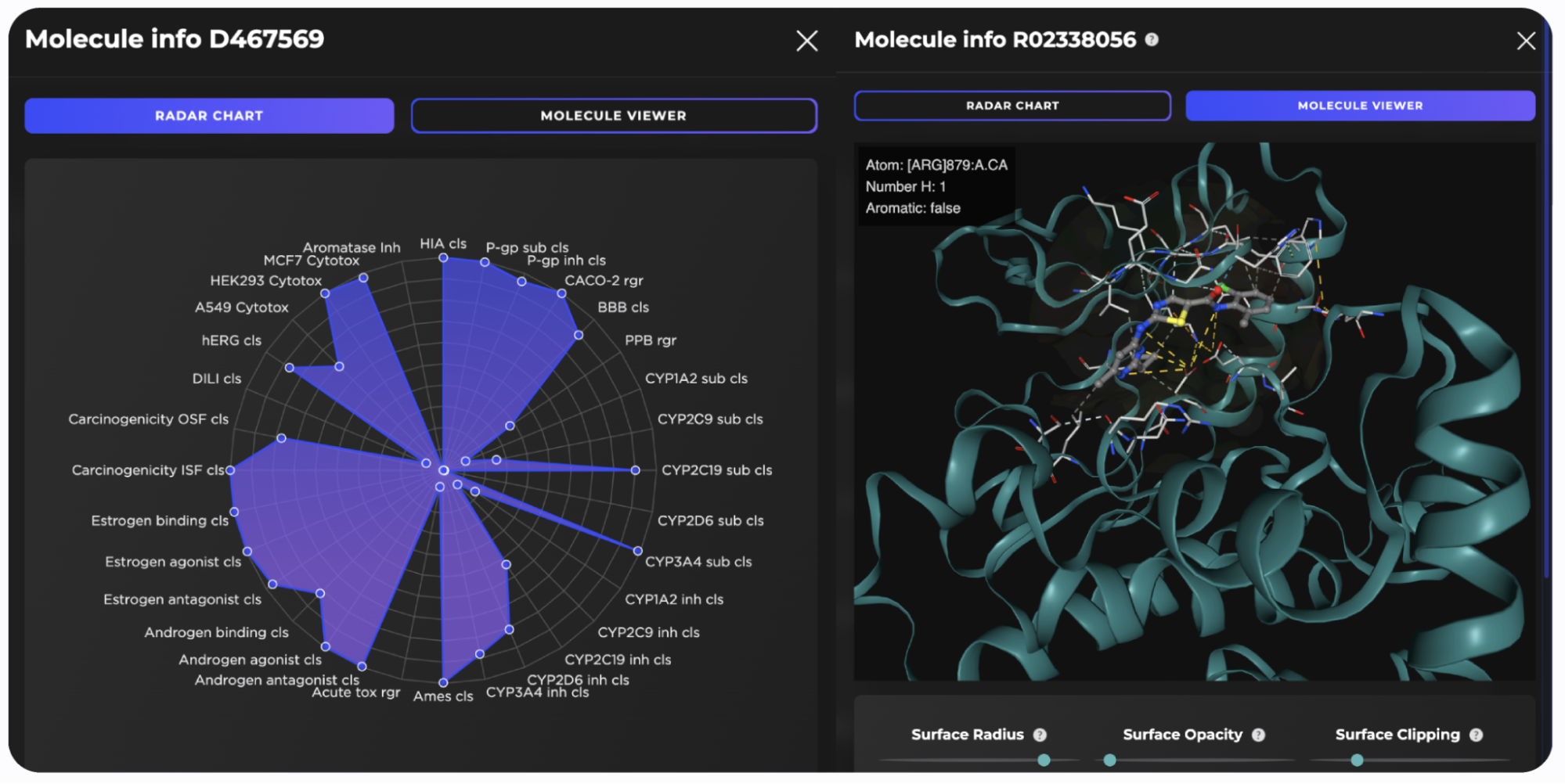
借助基础模型进行药物设计
Receptor.AI 平台使用户能够筛选数十亿种化合物,并根据具有高治疗潜力的知名目标蛋白评估筛选质量。通过将其平台的 AI 技术与 NVIDIA BioNeMo 云 API 相结合,Receptor.AI 能够简化蛋白质准备、大规模虚拟筛选和对接技术。
One Receptor.AI 的工作流程专注于理解长链多不饱和脂肪酸 (PUFA),这是癌细胞增殖的关键。考虑到人体氨基酸脱饱和酶 1 (FADS1,UniProt ID O60427) 在 PUFA 生物合成及其对癌症起源和生物学中的重要作用,该蛋白质被确定为有前景的药物靶点。在对 FADS1 进行选择性评估时,选择了三个同源氨基酸脱饱和酶域作为潜在的非标靶:FADS2 (ID O95864)、FADS3 (ID Q9Y5Q0) 和 FADS6 (ID Q8N9I5)。
Receptor.AI 精心策划了一个庞大的库,其中包含大约 43 亿种用于筛选的硅生成的小分子化合物,其中包括 25 种已知可与 FADS1 (称为配体)结合的化合物)这用于评估 Receptor.ai 的 AI 工作流程在处理化合物优先级和排序任务时的性能。重要的是,这些配体被排除在 AI 训练集之外,以保持其无偏测试方法的完整性。此外,Receptor.AI 整合了通过计算从已知 FADS1 配体衍生的分子,以展示 BioNeMo 和 Receptor.AI 平台强大的生成式 AI 功能。
用于筛选的预处理蛋白质和分子
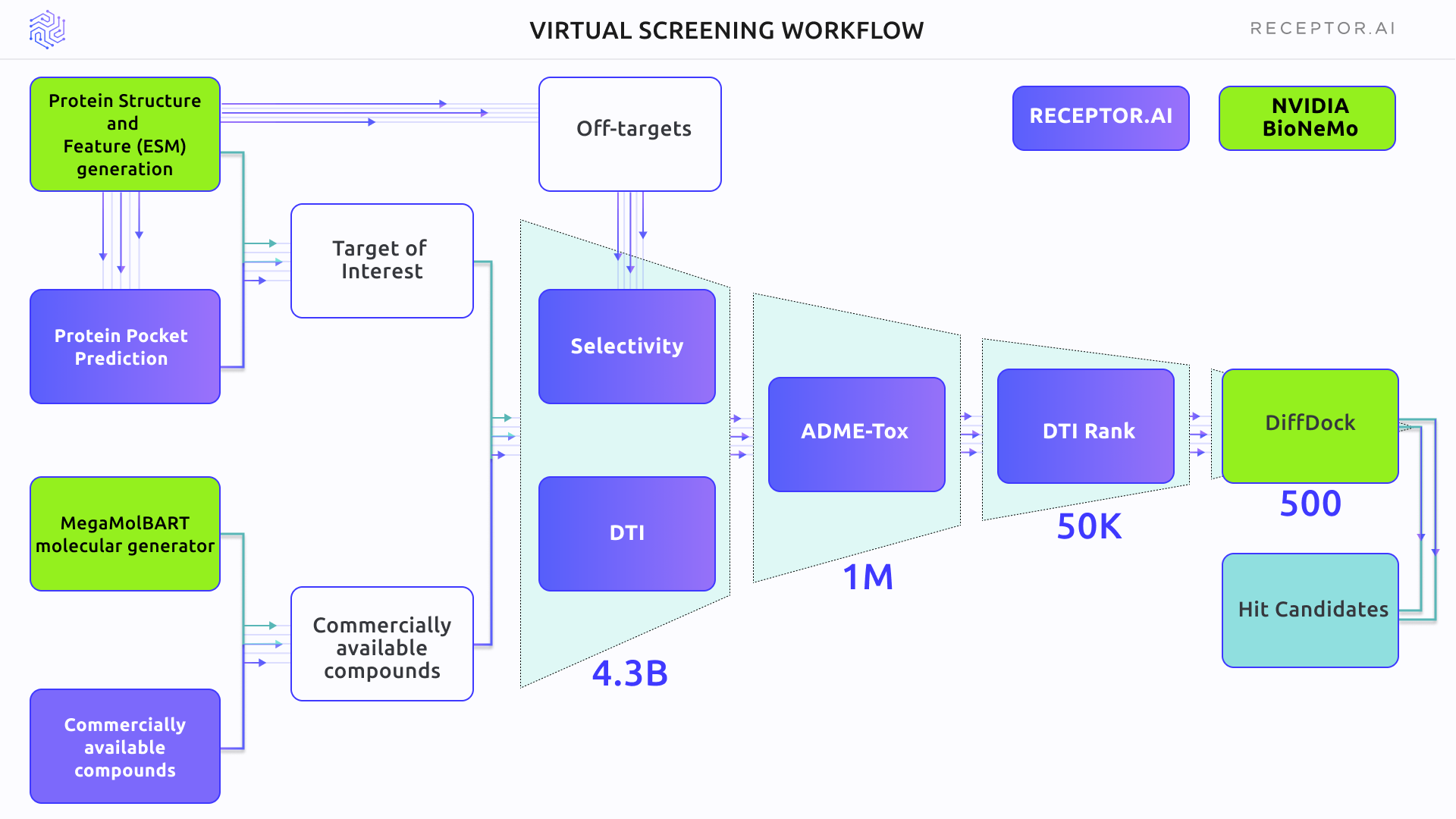
图 3 概述了虚拟筛选实验的方案。主要选择使用了 DTI 和选择性模型,然后是 ADME-Tox 评估。DTI 排名用于蛋白质组范围的选择性评估。使用 BioNemo DiffDock AI 对接获取入围化合物的结合姿势。在最后阶段,使用 Receptor.AI 共识函数对顶部候选分子进行排名。
蛋白质制备
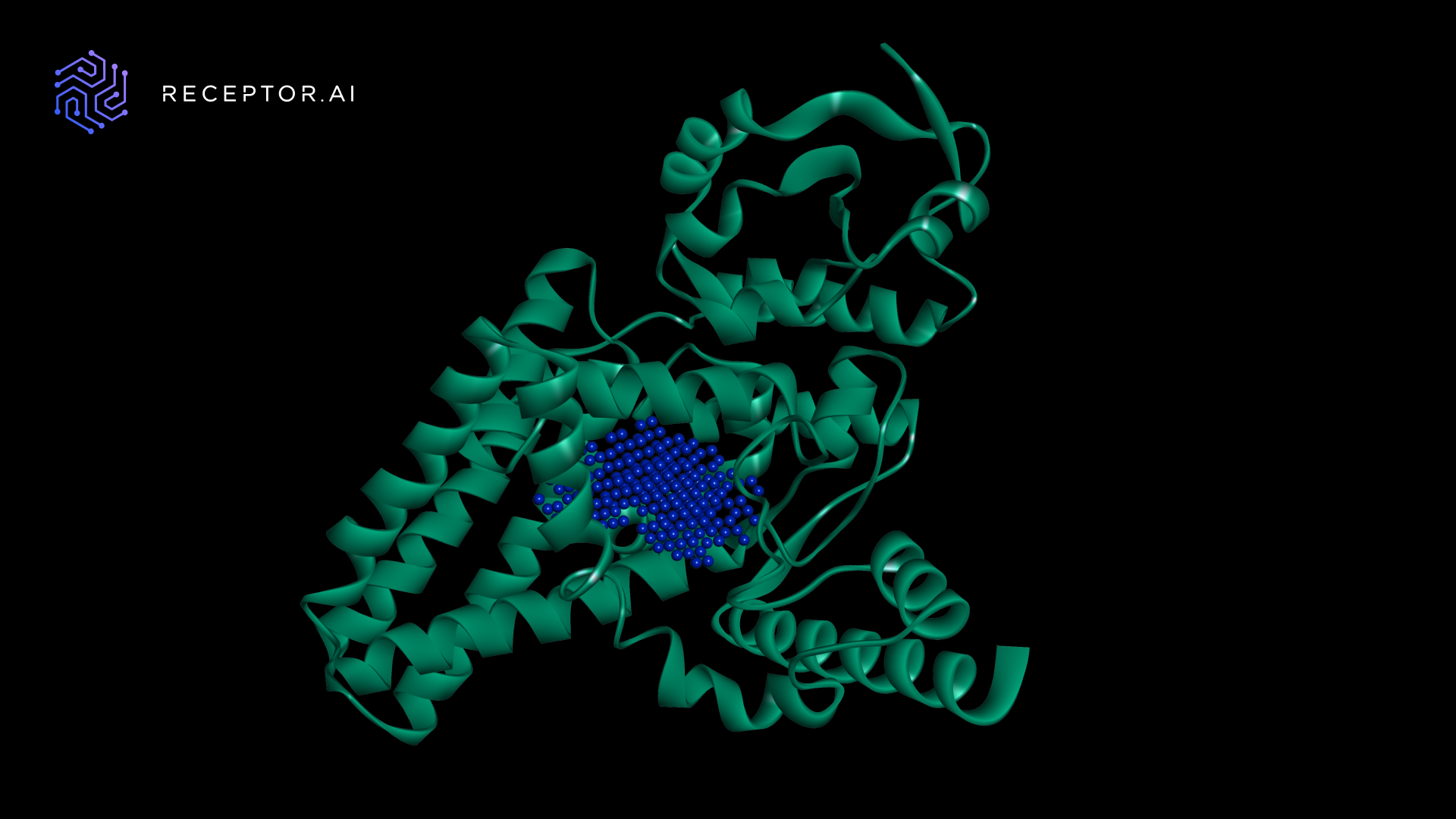
FADS1 蛋白没有可用的实验性 3D 结构,这是药物研发活动的基础。虽然其预测结构存在于 AlphaFold 蛋白质结构数据库,BioNeMo 通过 BioNeMo Cloud API (图 4) 在大约 17 分钟内对其进行了重新计算,具有很高的置信度。在处理新发现或突变的蛋白质时,此类功能至关重要。有关更多详细信息,请参阅 使用 AlphaFold 实现高度准确的蛋白质结构预测。
配体结合袋由PUResNet模型在NVIDIA Tesla T4 GPU 上进行AI 预测。根据InterPro FADS1 标注。
使用 BioNeMo Cloud API,计算了标和三种非标蛋白质的 ESM 编码。通过云 API 进行计算大约需要 1.4 秒,而使用 16 个虚拟 CPU 的 CPU 实例大约需要 6。0 秒。
筛选库准备
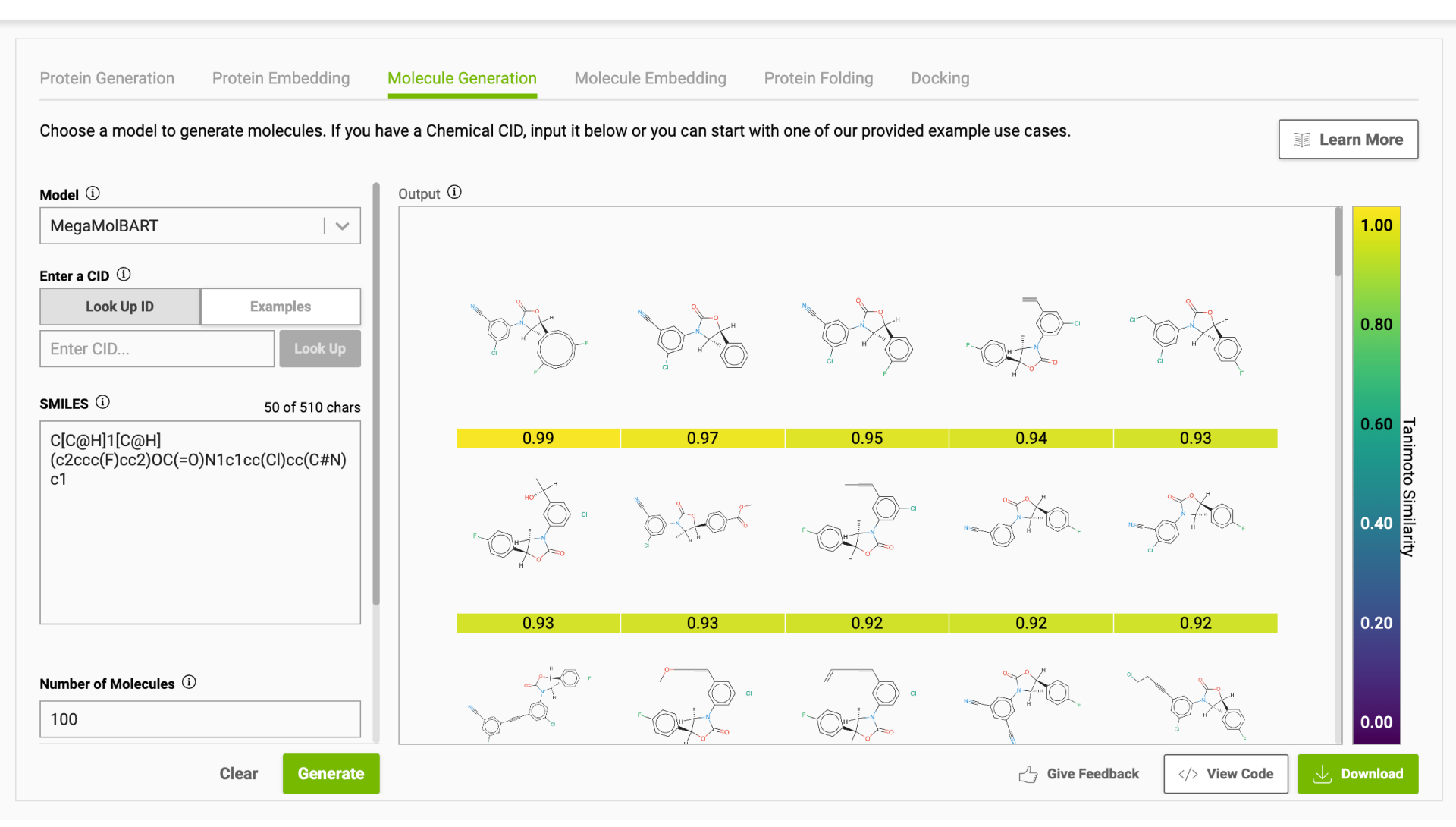
利用NVIDIA MegaMolBART模型,我们针对44种已知化合物,通过实验确定了FADS1的活性(IC50=1K nM)。这些数据来自ChEMBL数据库,我们据此生成了44万个新分子(每个已知化合物生成1万个新分子)。图5展示了生成的一些新分子示例。
Receptor.AI 将这些 44K 生成的化合物混合到 43 亿个虚拟库中。该库中的化合物可用作试验性阴性控制。他们还将 25 个随机选择的 FADS1 已知配体添加到筛选数据集,以评估其在最终结果中的排名。这样做的预期是,相当一部分已知配体会出现在最终排名候选名单之上。
GPU 驱动的虚拟筛选
Receptor.AI 命中识别管线包括 DTI、DTI 等级、选择性和 ADME-Tox 模块(图 6)。它用于将化学空间从大约 43 亿个化合物缩小到 500 个最有希望的候选化合物。筛选过程涉及 CPU 密集型和 GPU 密集型任务。表 1 比较了单个 NVIDIA T4 GPU 与具有 16 个 vCPU 的实例在虚拟筛选管线的不同阶段的性能。根据任务的不同,使用 GPU 可持续地将性能提升 1.1 倍到 11.3 倍。
| 技术 | 分子* | CPU 运行时间(小时) | GPU 运行时间(小时) | 加速系数 |
| 贸易和工业部 | 43 亿 | 33.9% | 11.3 | 3 |
| 选择性 | 1000 万 | 1 | 0.9 | 1.1 |
| ADME-Tox | 100 万 | 1.05 | 0.4 | 2.6 |
| DTI 排名(针对 9.3 K 种蛋白质) | 5 万 | 6.1 | 0.8 | 7.6 |
| 总计: | 42.05 | 13.4 | 3 | |
然后,Receptor.AI 平台识别出的前 500 种化合物会在没有有关键合袋位置的先验信息的情况下,与 FADS1 进行盲目对接。此过程使用了用于 DiffDock 的 BioNeMo 云 API。
在蛋白质准备过程中,使用 BioNeMo AlphaFold 模块在不到 17 分钟的时间内就可以自信地预测 FADS1 蛋白质结构。PUResNet AI 模型确定了配体结合袋。
在筛选库准备期间,BioNeMo Cloud API 可以更快地计算标和三个非标的 ESM 编码。该 API 由 BioNeMo Cloud 提供的相同技术支持,NVIDIA DGX 云与虚拟 CPU 相比。
最后,Receptor.AI 使用 NVIDIA T4 GPU 完成识别流程,该流程涉及 CPU 密集型和 GPU 密集型任务。具体来说,与 16 个 vCPU 的实例相比,GPU 上 43 亿个化合物虚拟筛选库的推理时间大大缩短。 NVIDIA GPU 提供的这种技术优势显著缩短了处理时间,并提高了整个实验的效率。
成果
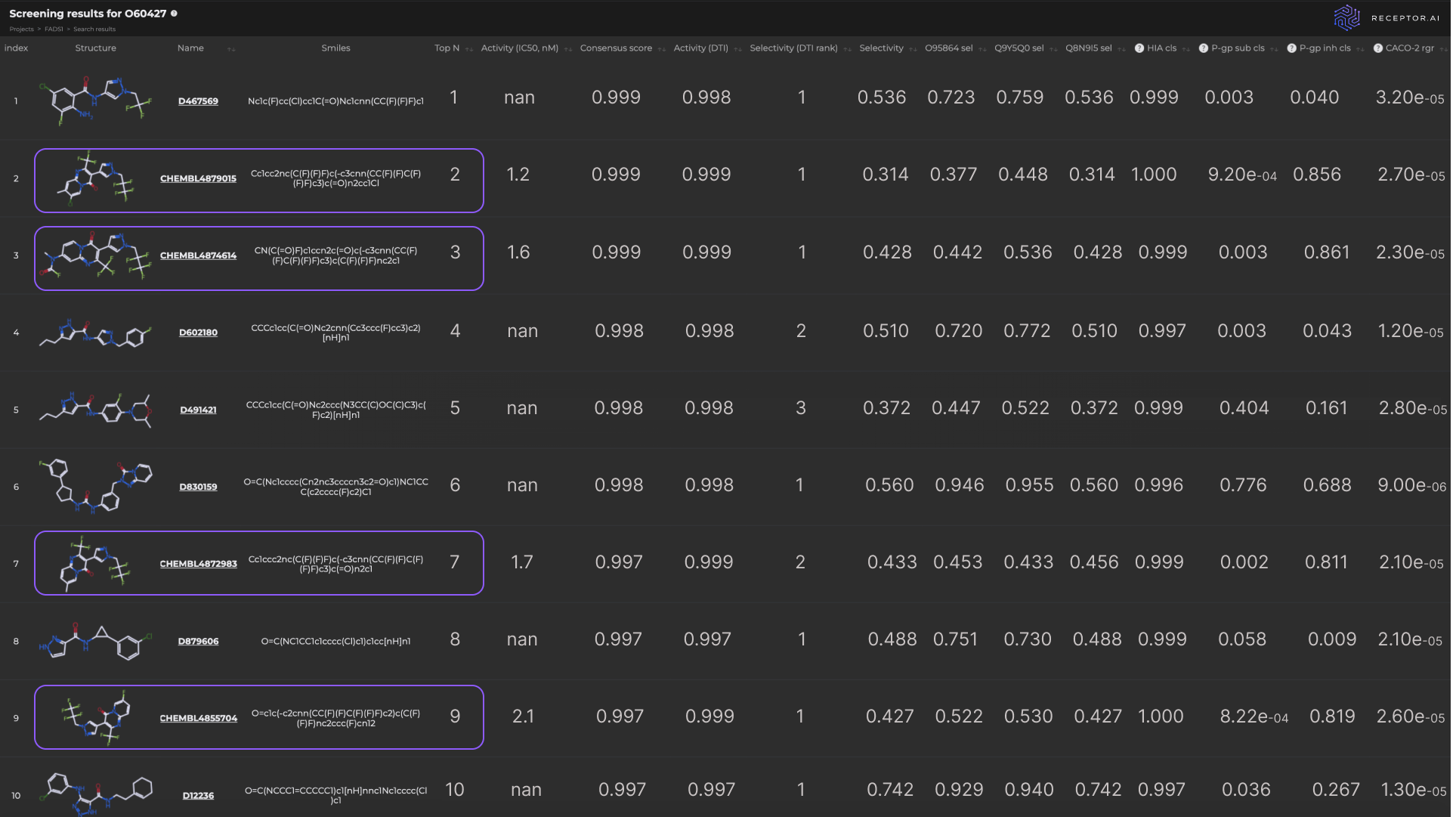
Receptor.AI 已从已知的 FADS1 配体中成功识别出几种命中化合物,以及与已知配体相比,与 FADS1 具有更高预测亲和力的新型化合物。在已知的 25 种 FADS1 配体中,有 4 种是在前 10 名命中候选配体中检测到的。在所有已知配体中,近一半(25 种配体中的 12 种)位于前 1K 中(图 7)。
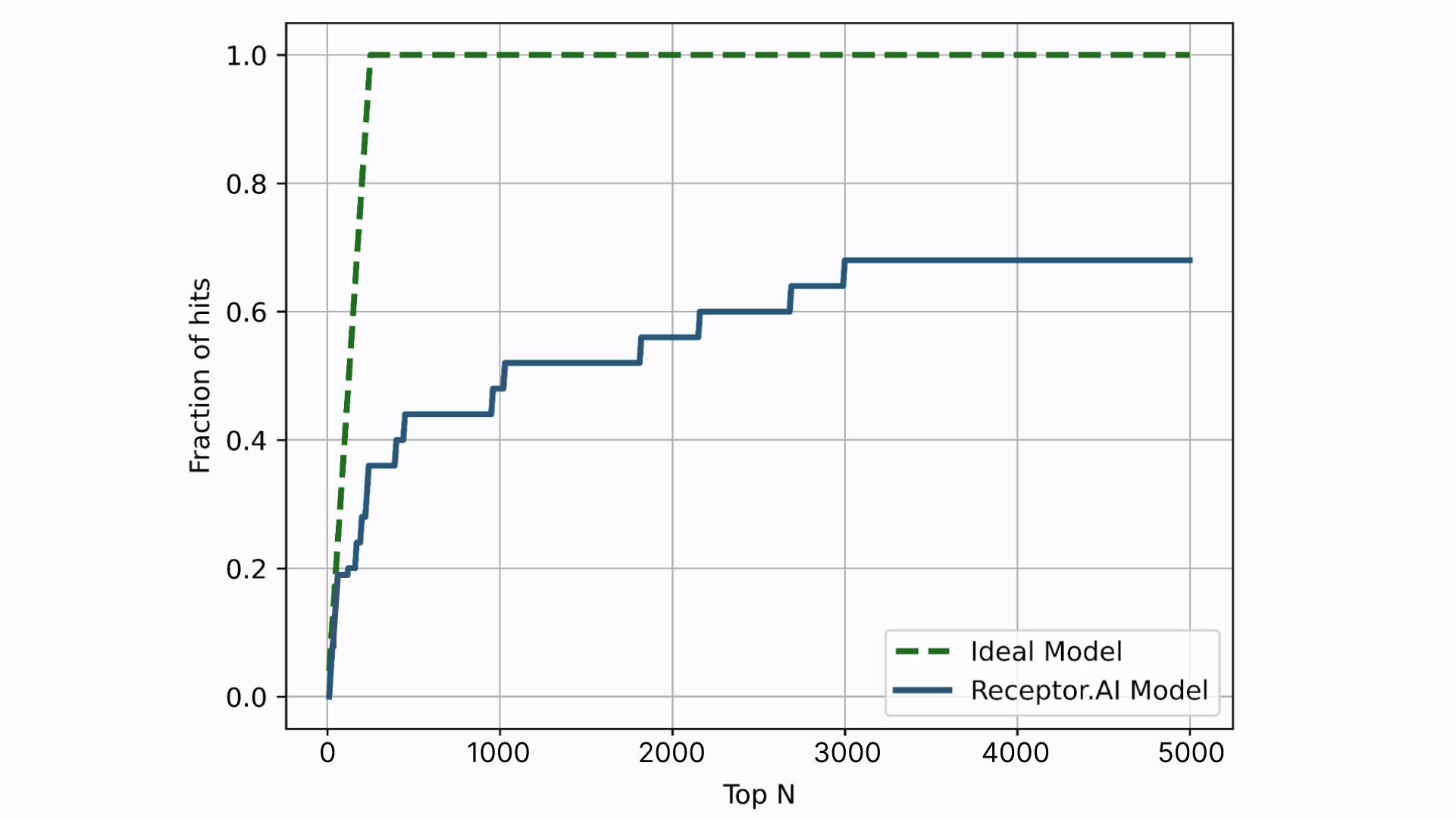
在最终排名列表中,位置 2、3、7 和 9 的分子是已知的具有高实验确定活度的配体。与已知配体相比,前 10 中的其他 6 种化合物对三个显式非目标具有更高的预测选择性(图 8)。前三个候选命中目标的预测 ADME-Tox 参数和对接姿势如图 9 所示。
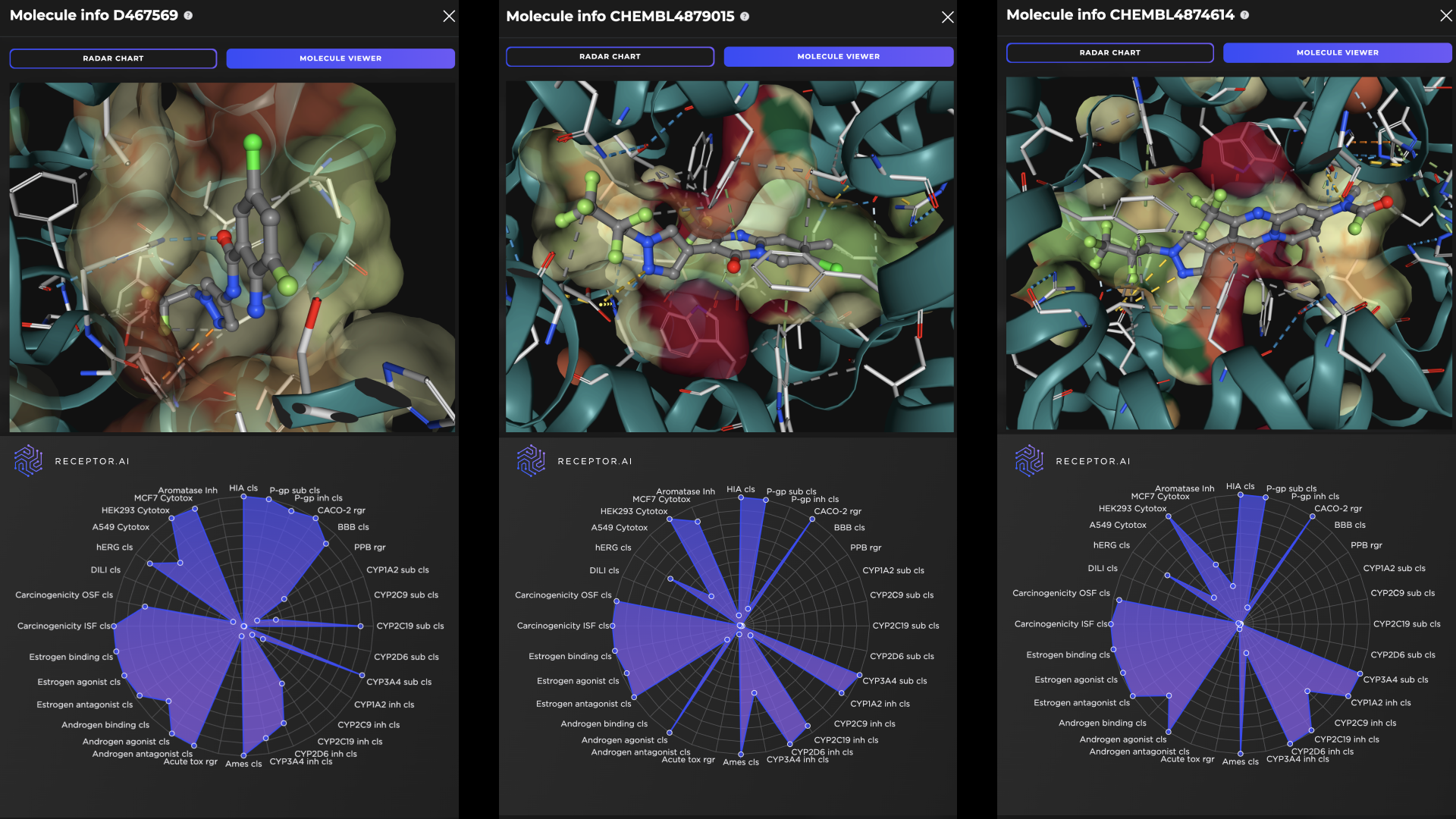
BioNeMo 云 API 提供的生化基础 AI 模型可以通过Receptor.AI用于在硅药物研发服务中的应用开发。这些模型在蛋白质结构预测、结合袋识别、蛋白质特征化、分子生成、虚拟化合物库筛选,以及基于 AI 的分子对接等方面表现出色。
包含 10 个预测候选项的最终列表包括 4 个经实验验证的目标蛋白质配体,这表明拟议的流程具有高度的实际相关性。
不仅在热门候选药物中成功识别了已知的配体,而且新的热门候选药物也出现了潜在的高活性和选择性。这表明拟议的流程在发现具有新型分子支架的潜在命中化合物方面的效用。
结束语
Receptor.ai 的一项重要发现是将加速计算集成到虚拟筛选流程中可节省成本。这一变化导致运行时间大幅减少,最终成本总体下降了 99%,从每实例小时 0.43 美元降至 0。22 美元。这展示了在大规模计算药物研发活动中利用加速计算的经济和效率优势,为行业和学术界提供了更经济高效、更快速的虚拟筛选模型。
接下来,我们将继续与 Receptor.AI 合作,利用 BioNeMo 框架和专有数据来训练 Receptor.AI 模型。我们的目标是基于 AI 技术,在高性能的 NVIDIA 加速工作站和云实例上运行分子动力学模拟,从而生成具有代表性的蛋白质构象集合。这一技术堆栈的进一步开发将致力于为生物技术和制药公司创建高效的药物研发平台。请访问 BioNeMo 框架 和 高级选择性工作流程 以获取更多信息。
深入了解加速 AI 驱动的药物研发,使用云 API 来利用强大、可定制的生成式 AI,而无需复杂的基础架构管理。开始使用 NVIDIA BioNeMo。