许多组织正在使用 NVIDIA Clara Parabricks 对大型人群项目、重症患者、临床工作流程和癌症基因组学项目进行快速人类基因组和外显子分析。他们的工作旨在准确、快速地识别致病变种,与加速的下一代测序以及加速的基因组分析保持同步。
最近, 8 月和 9 月的两份同行评审的科学出版物强调了 Clara Parabricks 在 de novo 和病原体工作流程中的速度、准确性和成本节约。
追踪疟疾传播的基因组变异鉴定
普渡大学首席研究员乔瓦娜·卡皮博士和她的团队试图了解 Clara Parabricks 相对于疟疾社区用于变种鉴定的现有方法的性能,以跟踪疟疾传播并使用 1000 个疟疾基因组监测抗疟药物耐药性。
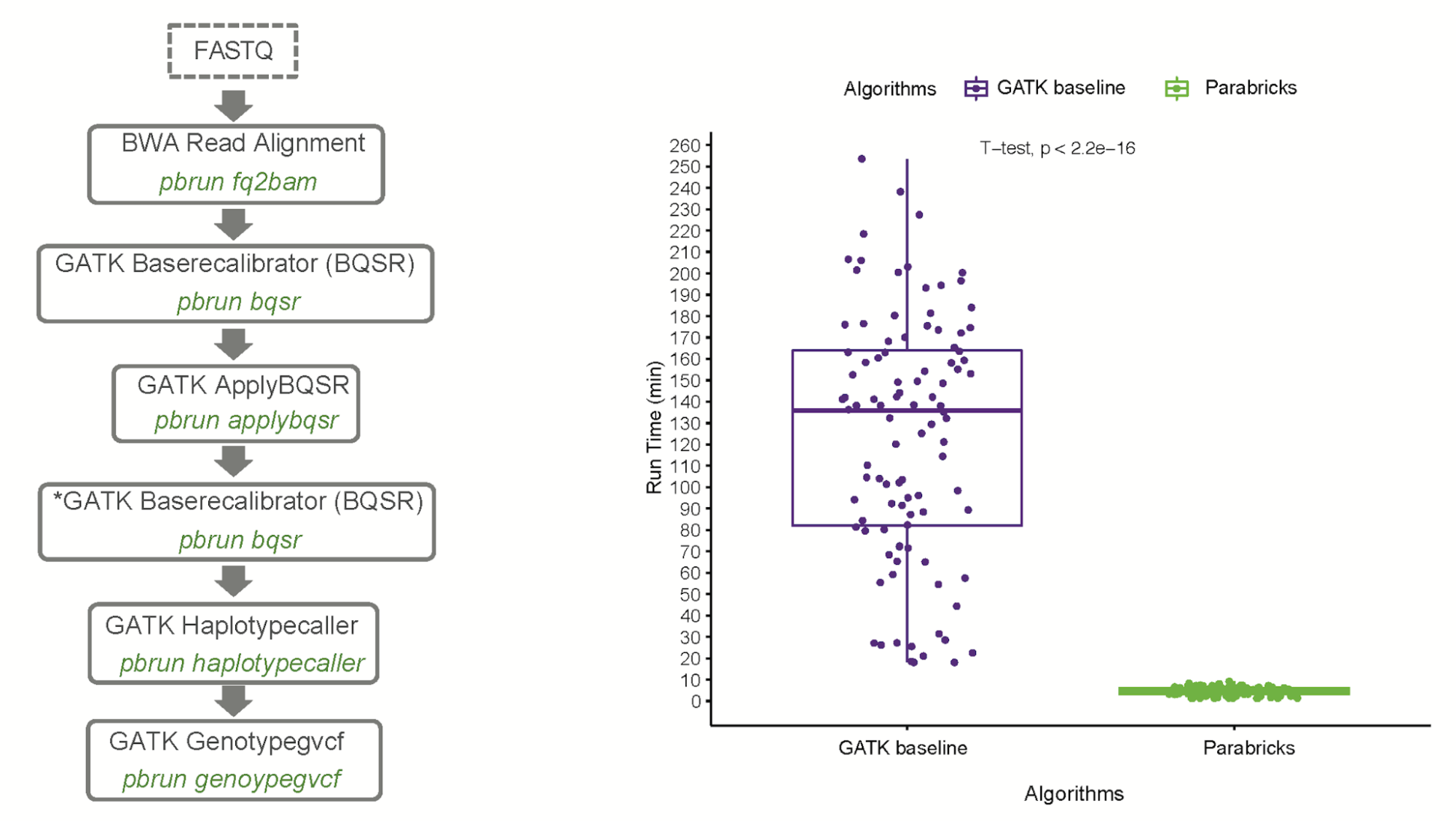
多年来一直在研究病原体基因组学的 Carpi 博士证明,与 CPU 传统管道相比,分析速度提高了 27 倍,成本降低了 5 倍,同时准确率达到 99.9% 。疟疾基因组相对较大( 24MB )且富含 AT ,这使得分析相当困难。 Carpi 博士使用了来自 MalariaGEN 财团的公开数据,这些数据是 Illumina 的原始数据。该研究发表在 A GPU-Accelerated Compute Framework for Pathogen Genomic Variant Identification to Aid Genomic Epidemiology of Infectious Disease: A Malaria Case Study 中,发表在 Briefings in Bioinformatics 中。
快速测序和分析全基因组病原体的能力有助于公共卫生官员了解疾病的传播、耐药性以及新变种的传播性和严重性。世界卫生组织( WHO )报告 241 million cases of malaria in 2020 相比 2019 年的 2.27 亿例, 2020 年估计有 627000 人死亡,比前一年增加 69000 人。
疟疾是由 Plasmodium 寄生虫引起的,这些寄生虫通过受感染的雌性 Anopheles 蚊子叮咬传播给人。非洲在全球疟疾负担中所占比例过高,五岁以下儿童占该地区总死亡人数的 80% 。
Carpi 博士指出,“能够在不到五分钟的时间内以 99.9% 以上的准确率以较低的成本为大规模全基因组 Plasmodium 研究生成分析就绪的变体输出,显著减少了大多数疟疾基因组学项目目前面临的计算瓶颈,并促进了流行国家的分散生物信息学分析。“访问 GitHub 上的 malaria-parabricks-pipeline 下载此 Clara Parabricks 疟疾工作流并了解更多信息。
在自闭症患者中发现 de novo 变异
另外,来自圣路易斯华盛顿大学的 Tychele Turner 博士和她的团队开发了一种快速基因组学工作流程,用于使用 GPU 加速的 Clara Parabricks 在自闭症患者中发现 de novo 变异( DNVs )。特纳博士是一位遗传学家/基因组学家,对理解人类疾病的遗传结构有着浓厚的兴趣。她的实验室专注于神经发育障碍的基因组学、基因组工作流程的优化以及应用新的基因组技术来理解疾病。这项研究发表在 De Novo Variant Calling Identifies Cancer Mutation Signatures in the 1000 Genomes Project 中,发表于 Human Mutation.
Turner 博士与 NVIDIA 基因组团队密切合作,将她的三重分析整合到 NVIDIA Clara Parabricks 中。特纳博士惊讶地看到,使用 NVIDIA Clara Parabricks 进行三重分析的周转时间加快了 100 倍。在 GPU 上生成 DNV 的初始分析使用只有 4 个 GPU 的服务器需要 8.5 小时,而在 CPU 上需要 800 小时。当团队在 GPU 上进一步并行化工作流程时,运行时间进一步缩短到不足一小时。
特纳博士的大部分职业生涯都集中在 DNV 上,这是一种存在于儿童 DNA 中但不存在于父母 DNA 中的新变种。这些 DNV 可以通过对孩子和父母的 DNA 进行测序,然后进行比较分析(称为三重分析)来评估。在普通人群中,每个个体约有 40 至 100 个 DNV ,大多数 DNV 不影响基因。
然而,当碱基对( a 、 T 、 C 、 G )中的单核苷酸变异( SNV )、小插入/缺失( indel )或结构变异( SV )改变基因并影响产生的蛋白质生产或功能时,通常会导致遗传病。一些神经发育障碍就是这样,在包括自闭症、癫痫、智力残疾和先天性心脏缺陷在内的表型中,患者体内蛋白质编码 DNV 的富集已被确认。
这些快速的结果不仅为科学发现带来了希望,也为特纳博士对当天临床结果的展望带来了希望。为了确认基于 GPU 的新工作流程中 de novo 变体调用的准确性,该团队利用 NVIDIA Clara Parabricks 研究了一个具有相同 DNA 的单卵孪生(也称为同卵孪生)的家庭。
结果显示,在基于 GPU 和以前基于 CPU 的工作流中, DNV 的数量相同,在这两种情况下都发现了大约 20% 的 CpG 站点,这表明 NVIDIA Clara Parabricks 工作流产生了相同的结果,但速度快了 100 倍。这意味着他们的自闭症基因组研究可以更快地完成,变异可以更快地被发现,并且有望更快地理解患者的见解。
特纳博士表示,“ GPU 的利用使快速生物信息学分析能够推进到一小时的基因组检查。”
利用新的基于 GPU 的 DNV 基因组分析工作流程,该团队继续研究来自 1000 Genomes Project 的序列数据,这是一个国际研究联合体,对来自非洲、东亚、南亚和欧洲人群的代表性队列进行了测序。 1000 基因组项目旨在通过对来自世界各地 26 个群体的 2600 名个体进行测序,描述和表征人类基因组中发现的变异,作为研究遗传多态性和表型之间关系的基础。
最近,纽约基因组中心对这些个体进行了深度测序,并公开了数据。该人群包括 602 个没有自闭症的家庭。这是第一次有机会将没有已知表型的 DNV 作为对照,以了解人群中 DNV 的水平,并将其与自闭症队列进行比较。
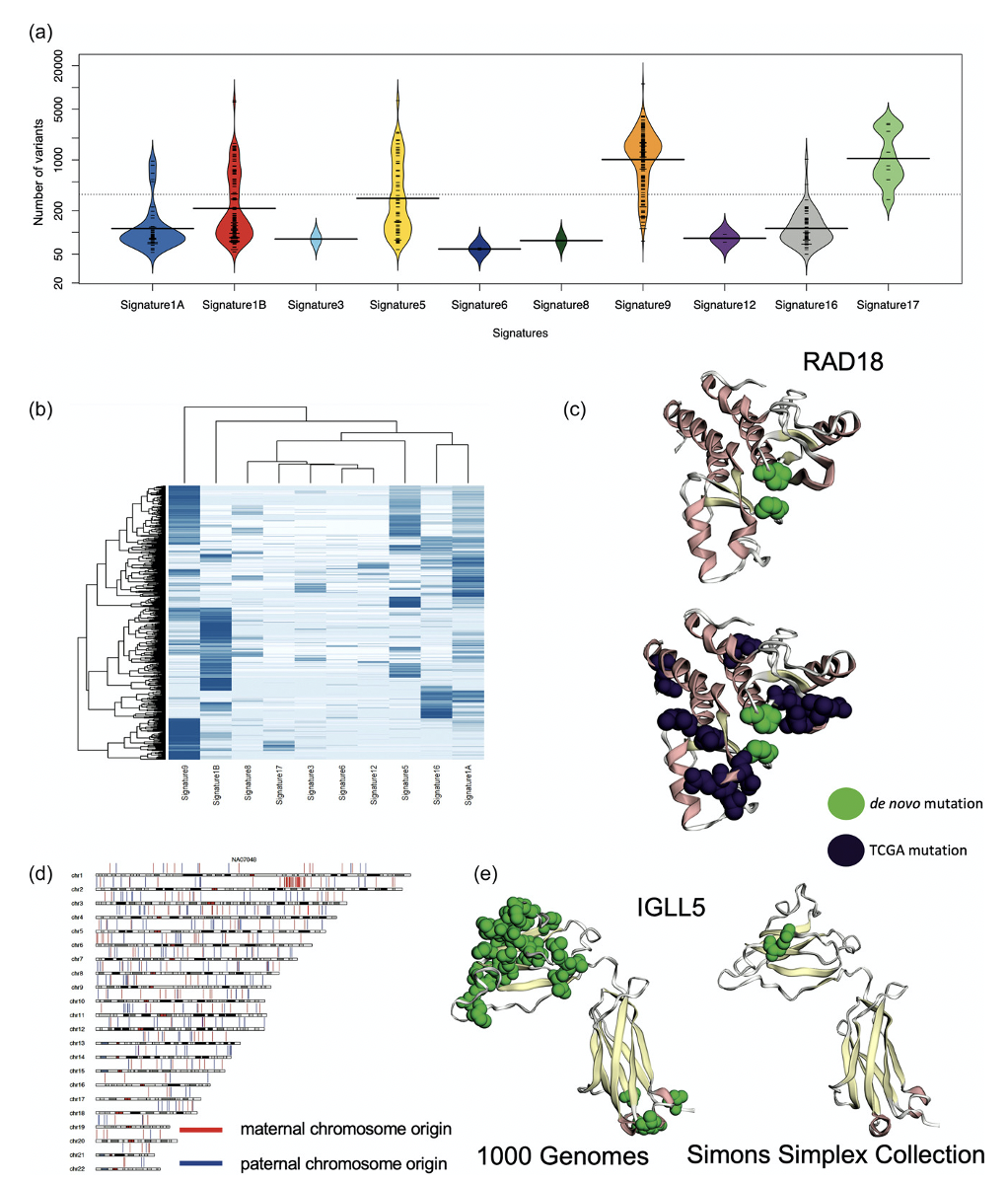
对 1000 名基因组计划个体的 DNV 分析最终让特纳博士的团队感到惊讶。他们发现 DNV 数量呈双峰分布,峰值为 200 ,略大于预期,峰值为 2000 ,远大于预期。特纳博士查看了 1000 基因组计划数据中的各个队列,并注意到 CEU 人群是欧洲个体的队列,研究时间更长,因此也培养了更多,可能导致更多的细胞系伪影。
在队列中,有一名被确定为 NA12878 的个体进行了多次测序: 2012 年、 2013 年、 2018 年和 2020 年进行了两次测序。特纳博士表明, DNV 随着时间的推移而增加。 2020 年的 DNV 最多,支持了 2020 年样本中的细胞系伪影比 2012 年样本更多的结论。该团队得出结论,尽管 1000 基因组项目是基因组研究的一个极好的数据来源,但由于细胞系伪影的普遍存在,它可能不适合筛选患者对照的数据集。
尽管 1000 基因组计划提供了关键的生物学和实践见解,但只有 20% 的儿童拥有预期数量的 DNV ,大量证据表明过量的 DNV 是细胞系伪影。过量的 DNV 与 B 细胞淋巴瘤癌的突变特征相匹配,表明细胞系伪影不是以随机方式累积的。
蛋白质编码的 DNV 在 DNA 修复基因中被鉴定,并可能导致过量的 DNV 。 602 名个体的队列对于已知在 B 细胞淋巴瘤中具有过量突变的 IGLL5 中的蛋白质编码 DNV 具有重要意义,并且具有这些 DNV 的个体都具有大于 100 个 DNV 。蛋白质编码 DNV 在临床相关的变异位点中被识别,在使用该数据作为患者的二进制过滤集时需要谨慎。未来进行基因组测序的基因组研究应侧重于基于家族的方法或利用直接从血液中提取的 DNA 建立良好的对照和参考数据库。
特纳博士评论道:“我的实验室很高兴开发出一种 de novo 变体调用工作流,该工作流利用 GPU ,使我们能够快速分析近 4800 个全基因组测序的亲子三人组,以获得重要的生物学见解。”
为基因组研究提供动力的一套加速工具
Clara Parabricks v4.0 是一个比以前版本更专注的基因组分析工具集,具有快速比对、金标准处理和高精度变体调用。它提供了自由无缝地交织 GPU 和 CPU 任务的灵活性,并优先考虑基因组学工作流程中最流行和瓶颈工具的 GPU 加速。 Clara Parabricks 还可以整合基因组学中的前沿深度学习方法。
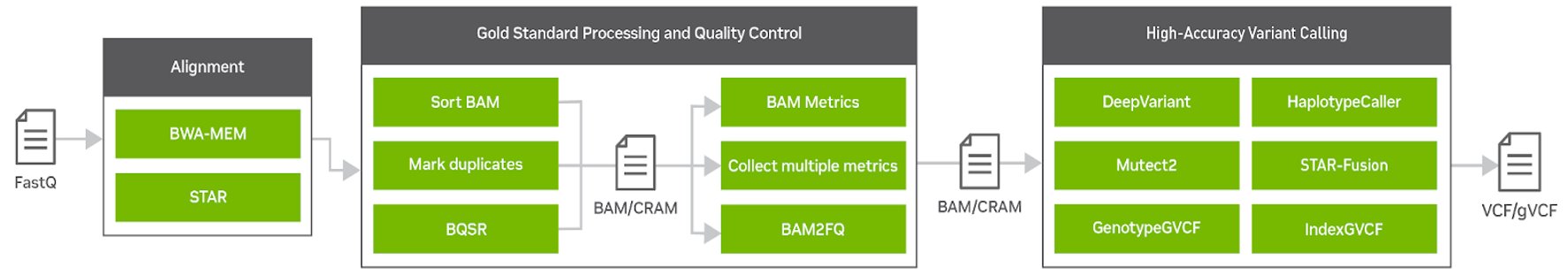
您可以免费注册 下载 Clara Parabricks 。您还可以请求一个免费的 Clara Parabricks NVIDIA LaunchPad Lab 演示,体验用于外显子组和全基因组数据集的种系和体细胞分析的加速行业标准工具。
有关 Clara Parabricks 的更多信息,包括可用工具的技术细节,请查看 Clara Parabricks 文档 。