训练拥有数十亿乃至数万亿参数的模型依赖于先进的并行计算技术。研究人员需要在不牺牲计算速度和内存效率的前提下,合理组合并行策略,选用高效加速库,并集成FP8、FP4等低精度计算格式。
加速框架虽有一定帮助,但适应这些特定方法可能会显著拖慢研发进度,因为用户通常需要学习一套全新的代码库。
NVIDIA BioNeMo Recipes 通过提供基于 PyTorch 和 Hugging Face(HF)等常用框架的分步指南,降低了大规模模型训练的入门门槛,简化并加速了整个流程。我们展示了如何集成 NVIDIA Transformer 引擎(TE)等加速库,结合全分片数据并行(FSDP)和上下文并行等技术,有效提升训练速度与内存效率,并增强可扩展性。
在这篇博客文章中,我们展示了如何利用 Hugging Face 的 ESM-2 蛋白质语言模型,并结合原生 PyTorch 训练循环,加速基于 Transformer 的生物AI模型的训练过程。
- 借助 TE 实现加速,
- 集成 FSDP2 以支持自动并行,
- 采用 Showin 序列打包技术提升性能。
您只需使用 PyTorch、NVIDIA CUDA 12.8 以及以下资源,即可快速上手。
将 Transformer 引擎集成到 ESM-2 中
TE 通过优化 Transformer 计算(特别是在 NVIDIA GPU 上)实现了显著的性能提升。它可轻松集成到现有的训练流程中,无需对数据集、数据加载器或训练器进行大规模修改。本节将借鉴 BioNeMo 配方的设计思路,展示如何将 TE 成功整合到 ESM-2 等模型中。
在大多数用例中,使用来自 TE 的现成 TransformerLayer 模块非常简便。该模块将所有融合的 TE 操作和最佳实践封装在一个内建组件中,有效减少了冗余代码和配置工作。以下代码片段展示了如何在 ESM-2 中集成 TE。完整实现可参考 bionemo-recipes 中 NVEsmEncoder 的类定义。
import torch
import transformer_engine.pytorch as te
from transformer_engine.common.recipe import Format, DelayedScaling
class MyEsmEncoder(torch.nn.Module):
def __init__(self, num_layers, hidden_size, ffn_hidden_size, num_heads):
super().__init__()
self.layers = torch.nn.ModuleList([
te.TransformerLayer(
hidden_size=hidden_size,
ffn_hidden_size=ffn_hidden_size,
num_attention_heads=num_heads,
layer_type="encoder",
self_attn_mask_type="padding",
attn_input_format="bshd", # or 'thd', read below.
window_size=(-1, -1), # disable windowed attention
) for _ in range(num_layers)
])
# Optionally add embedding, head, etc.
def forward(self, x, attention_mask=None):
for layer in self.layers:
x = layer(x, attention_mask=attention_mask)
return x
# Layer configuration
layer_num = 8
hidden_size = 4096
sequence_length = 2048
batch_size = 4
ffn_hidden_size = 16384
num_attention_heads = 32
dtype = torch.bfloat16
# Synthetic data (batch, seq, hidden) for bshd format
x = torch.rand(batch_size, sequence_length, hidden_size).cuda().to(dtype=dtype)
attention_mask = torch.ones(batch_size, 1, 1, sequence_length, dtype=torch.bool).cuda()
myEsm = MyEsmEncoder(layer_num, hidden_size, ffn_hidden_size, num_attention_heads)
myEsm.to(dtype=dtype).cuda()
fp8_recipe = DelayedScaling(fp8_format=Format.HYBRID)
with te.fp8_autocast(enabled=True, fp8_recipe=fp8_recipe):
y = myEsm(x, attention_mask=attention_mask)
如果您的架构与标准 Transformer 模块不同,仍然可以在层级上集成 TE。其核心思想是将标准 PyTorch 模块(例如 nn.Linear、nn.LayerNorm)替换为对应的 TE 模块,并通过 FP8 自动类型转换来获得显著的性能提升。TE 为常见层(如 Linear 层、融合的 LayerNormLinear 层)以及注意力模块(如 DotProductAttention 和 MultiheadAttention)提供了多种优化实现。有关支持模块的完整列表,请参考 TE 官方文档。
高效的序列打包
当样本具有不同序列长度时,标准输入数据格式可能效率较低。例如,在上下文长度为 1024 的 ESM-2 预训练中,填充 token 的比例可能高达约 60%,导致在不参与模型注意力计算的 token 上浪费大量计算资源。在模型内部,网络通常以四个维度表示输入序列的隐藏状态:批量大小(B)、最大序列长度(S)、注意力头数(H)和头部隐藏维度(D),即 BSHD 格式。<!–
作为替代,现代注意力内核支持用户在无需填充 token 的情况下提供打包后的输入,并通过索引向量来标识不同输入序列之间的边界。此时,隐藏状态以扁平化张量的形式表示,其维度为【扁平化输入 token 数量 (T)、注意力头数 (H)、每个头的隐藏维度 (D)】,即 THD。如图 1 所示,通过移除填充 token(灰色部分),该方法不仅减少了显存占用,还提升了 token 处理吞吐量。
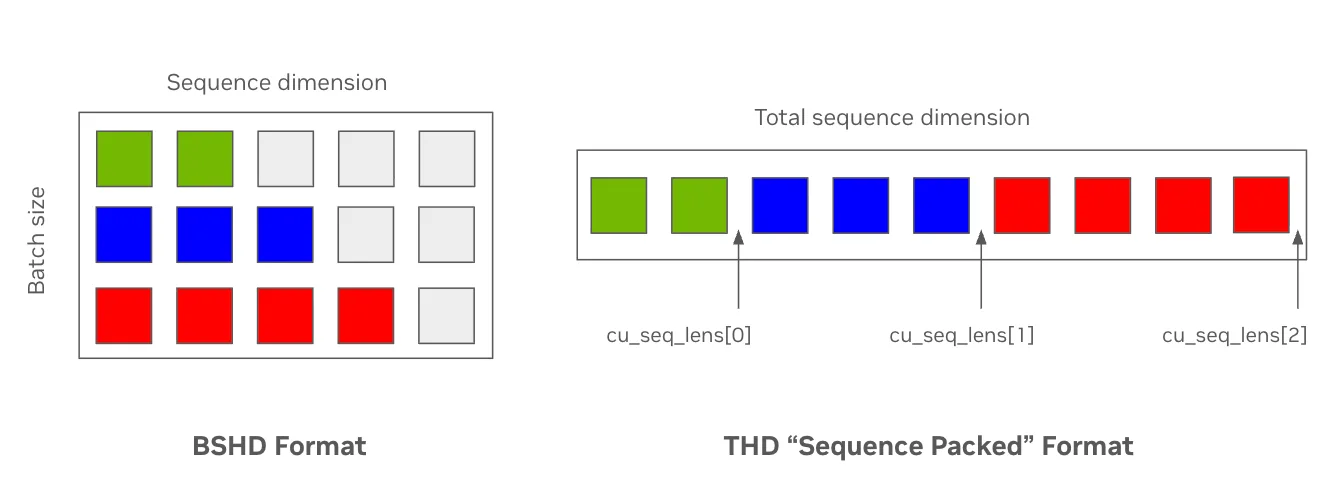
通过在相关层中添加 attn_input_format 参数,并支持标准的闪存注意力机制中的累积序列长度关键字参数(cu_seq_lens_q)),TE 使得这一优化过程相对简便。这些数据可通过支持 THD 感知的整理器(例如 Hugging Face 的 DataCollatorWithFlattening)或 BioNeMo Recipes 中实现的带掩码版本生成。
def sequence_pack(input_ids, labels):
# input_ids is a list of sequences: [(S1,), (S2,), ..., (SN,)] of shape (B,S)
# Flatten and track sequence boundaries
# Determine the length of each sequence
sample_lengths = [len(sample) for sample in input_ids]
# Flatten the input_ids and labels
flat_input_ids = [token for sample in input_ids for token in sample]
flat_labels = [label for sample in labels for label in sample]
# Create a list of cumulative sums showing where the sequences start/stop
# Note: for self attention cu_seqlens_q and cu_seqlens_kv will be the same
cu_seqlens = torch.cumsum(torch.tensor([0] + sample_lengths), dim=0, dtype=torch.int32)
max_length = max(sample_lengths)
return {
"input_ids": torch.tensor(flat_input_ids, dtype=torch.int64),
"labels": torch.tensor(flat_labels, dtype=torch.int64),
# These are the same kwargs used by `flash_attn_varlen_func`, etc.
"cu_seqlens_q": cu_seqlens,
"cu_seqlens_kv": cu_seqlens,
"max_length_q": max_length,
"max_length_kv": max_length,
}
TE 和序列封装开/ 关性能
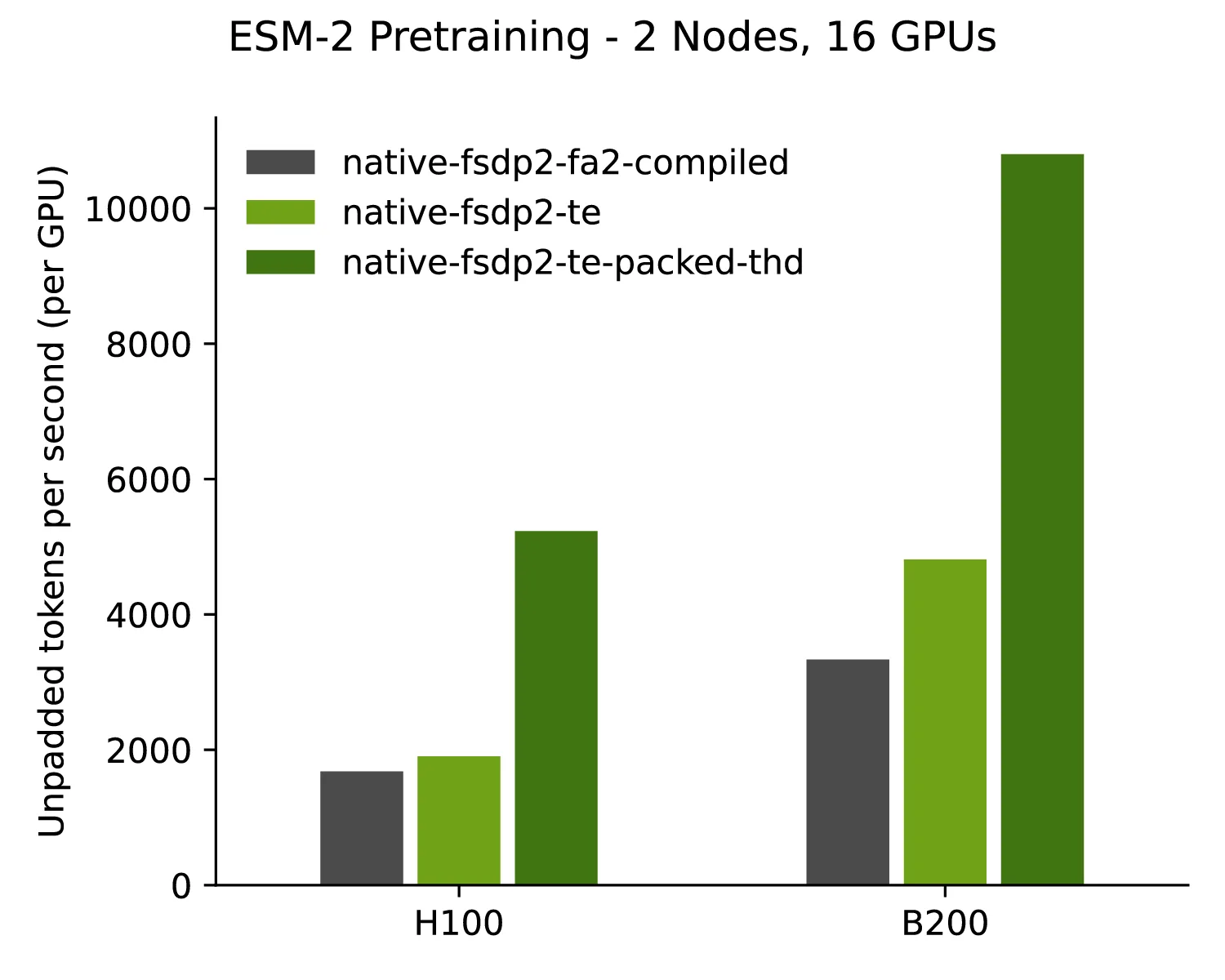
图2展示了采用TE后token吞吐量显著提升的性能对比,表明TE能够更充分地提升NVIDIA GPU的计算效率。
EvolutionaryScale 在其新一代模型中集成了 Transformer 引擎。
“ESM3 是目前基于生物数据训练的规模最大的基础模型之一。在训练这一拥有 98 亿参数的模型时,集成 NVIDIA Transformer 引擎对于实现高吞吐量和高效 GPU 利用率至关重要。”EvolutionaryScale 联合创始人兼工程副总裁 Tom Sercu 表示,“FP8 所带来的精度与速度提升,结合融合层的优化内核,使我们能够在多块 NVIDIA GPU 上突破计算与模型规模的限制,助力科学界借助我们的前沿模型更深入地探索生物学奥秘。”
Hugging Face 互操作性
TE 的一大优势在于其与现有机器学习生态系统的良好互操作性,包括 Hugging Face 等广受欢迎的库。这意味着,即使在使用 Hugging Face Transformers 库加载的模型时,您依然能够充分发挥 TE 带来的性能优势。
TE 层可直接嵌入 Hugging Face Transformer PreTrainedModel,并完全兼容 AutoModel.from_pretrained。如需获取预优化模型,可访问 NVIDIA BioNeMo 系列 on the Hugging Face Hub。
该过程通常包括加载 Hugging Face 模型,然后准确识别其中的标准 PyTorch 层(例如 nn.Linear、nn.LayerNorm 和 nn.MultiheadAttention),并将其替换为经过 TE 优化的对应层。为确保 TE 层能够正确集成到模型的前向传播过程中,通常需要对部分层进行重命名,或使用自定义的模型包装器。<!–
开始使用
BioNeMo Recipes 致力于为基础模型构建者提供高效的加速与扩展能力。为了帮助我们打造更强大、更实用的工具包,我们诚挚期待您的反馈。欢迎尝试我们的方法,并通过提交拉取请求或在 GitHub 上提出问题来参与贡献。