作为一名对医疗应用人工智能感到兴奋的本科生,我很高兴能加入 NVIDIA Clara 部署团队进行实习。这是一个完美的组合:有机会在一家领先的技术公司工作,加速和采用人工智能,同时为未来(和现在)的团队建设做出贡献用于医疗保健的人工智能部署。接下来的几个月充满了从才华横溢但谦逊的同事那里学到的东西,学习了 CUDA 编程等新技能,以及关注组织病理学数据带来的独特技术挑战的机会。
什么是 Clara Deploy?
Clara Deploy SDK 是一个基于容器的云本机开发和部署框架,用于智能医院中的多 AI 和多域工作流。它使您能够定义由多个阶段组成的基于容器的管道,每个阶段由操作员定义。管道由多个运算符组成,是从数据源到数据接收器的有向无环图( DAG )。每个操作符都是管道的一个步骤,比如加载输入、预处理、人工智能推理等等。
在我探索建立 NVIDIA Clara Deploy 平台和运行 AI 推理管道的过程中,我获得了部署 AI 工作流挑战的第一手经验,特别是在标准化工作流和扩展执行方面。在运行数字病理管道的过程中,我意识到了 I / O 和预处理步骤的性能瓶颈,这些步骤通常不会 GPU 加速。这影响了我在实习期间专注于加速数字病理学预处理过滤器的选择。
什么是 cuCIM ?
cuCIM 是一个 RAPIDS 库,用于加速 n 维图像处理和图像 I / O ,重点是医学成像应用。 cuCIM 由 I / O 、文件系统和操作模块组成。 cuCIM 中的操作可以使用插件架构进行扩展。 cuCIM 在医学图像处理应用方面处于领先地位,我很高兴在 NVIDIA 工作期间接触并参与其中。
项目动机
组织病理学分析数字化的一个重大挑战是病理学图像中观察到的染色变化。这些图像可能由多种因素引起 染色差异大 ,包括染色供应商、储存条件、染色协议、数字扫描仪等。
鉴于因素的范围,在图像采集期间控制染色变化是不切实际的。取而代之的是,一个称为染色标准化的图像预处理步骤通常用于算法标准化图像染色。染色规格化过滤器接受源图像和目标图像作为输入。将源图像进行染色归一化,目标图像包含理想染色,并将其转移到源图像。最终,标准化的源图像作为输出返回。
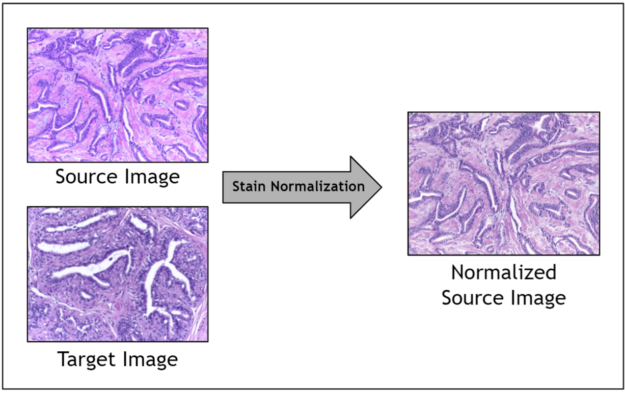
先前的工作表明,在数字病理 AI 管道中,作为预处理步骤的染色归一化可以缩短训练时间,提高准确性,并使来自不同来源的数据能够一起使用。由于染色病理图像的稀缺性,您在相对较小的数据范围内操作,因此染色标准化使您能够优化在噪声染色变化中获得的信号。
然而,之前的染色标准化实施相对缓慢,因为它们没有 GPU 加速。有机会实现 GPU 加速染色归一化算法,并为数字病理 AI 管道实现快速有效的预处理。
加速数字化病理学染色标准化
染色标准化方法分为三大类:
- 全局颜色归一化
- 色斑反褶积后的颜色归一化
- 基于深度网络的颜色传递
有关更多信息,请参阅 染色颜色自适应归一化( SCAN )算法:数字病理学中组织染色的分离和标准化 。
我选择专注于基于染色反褶积的方法,因为之前的文献显示,与基于深度网络的方法相比,与全局颜色归一化相比,其性能更高,并且在维持生物结构完整性方面具有更好的理论保证。
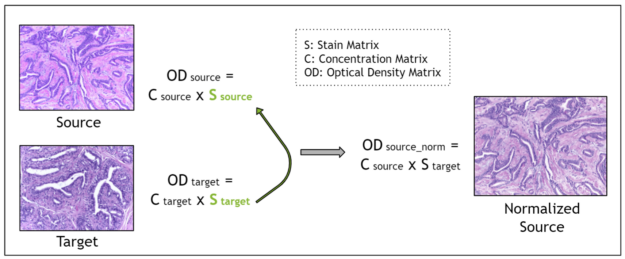
基于染色反褶积的方法假设每个图像都具有染色矩阵的特征,该矩阵包含 H & E 染色图像中两种染色(苏木精和曙红)的红、绿、蓝( RGB )值。
使用 比尔 – 兰伯特定律 将 RGB 图像转换为光密度图像。然后,光密度图像可以与该图像的像素浓度矩阵和染色矩阵的乘积相关。像素浓度矩阵表示每个像素的每个染色的浓度。如果使用 马森科法 估算染色矩阵,则可获得浓度矩阵。
最后,对于染色归一化,源图像的染色矩阵被替换为 目标图像的染色矩阵 。这用于将染色轮廓从目标图像传输到源图像。因为源图像的浓度矩阵不变,所以生物结构的形态保持不变。用于估计染色矩阵的 Macenko 方法是一种使用 奇异值分解 的无监督方法。
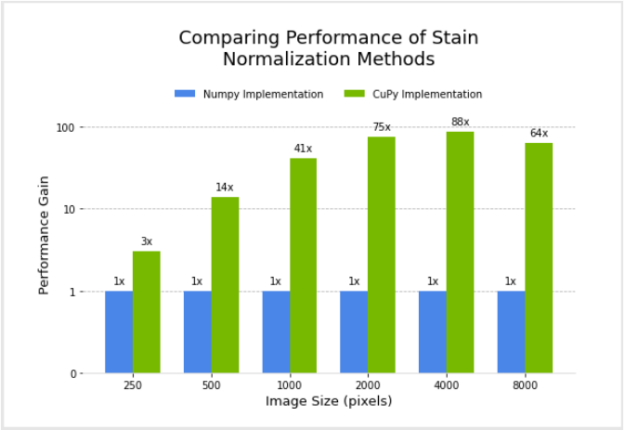
在修改了 NumPy 中的现有版本后,我为 CuPy 中的 Macenko 染色标准化方法设计并实现了一个过滤器。接下来,我比较了两者的性能。
图 3 显示了使用 NVIDIA DGX-1 对不同图像大小的 NumPy 和 CuPy 实现的着色归一化的相对性能。 CuPy 实现的性能根据相对于 NumPy 实现的加速因子绘制。
考虑到使 GPU 加速染色标准化能够用作数字病理学管道的预处理步骤的目标,我开始将该过滤器作为转换(基于阵列和基于字典)集成到 MONAI 。 MONAI 是一个基于 PyTorch 的开源框架,用于医学成像的深度学习。完全集成后,可以将染色归一化变换添加到 Clara Train 或 MONAI 中的病理管道中。
彩色转换滤波器的加速
接下来,我在 CUDA C ++中实现了颜色转换 RGB2HED 函数,这是一个常用的函数,在 scikit-image 和 Cuim-Python 层中可用。从 RGB 到 HED 的颜色空间转换与色斑归一化密切相关,因为该函数涉及获得色斑浓度值,假设色斑向量是一个恒定的、预先计算的近似值。这忽略了不同图像染色之间的差异。这个功能通过 C ++的操作符插件机制集成到 CuCIM 中。
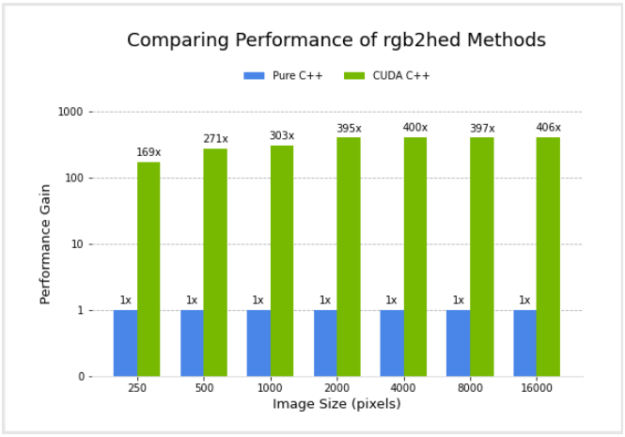
我比较了纯 C ++实现和 CUDA C ++实现的性能。图 4 显示了使用 NVIDIA GV100 GPU 和 Intel ( R ) Core ( TM ) i7-7800X CPU 的两个版本在不同图像大小下的相对性能。 CUDA C ++实现的性能以相对于纯 C ++实现的加速因子的形式绘制。
需要注意的是,性能提升并没有考虑到与 GPU 之间的任何数据传输。我之所以这样做,是因为我正在考虑一种常见的场景,即在图像处理工作流中的几个后续操作中,数据传输通过留在 GPU 上而最小化,并且只在最后发生向主机的传输。
结论
总之,我的实习项目的重点是加速数字病理学的颜色转换过滤器。具体来说,我致力于设计和实现 Macenko 染色归一化方法,使用 CuPy 进行 GPU – 加速。我开始将其集成到 MONAI 中,作为一种转换,以便将来用作数字病理管道的预处理步骤。接下来,我在 CUDA C ++中实现了颜色转换 RGB2HED 函数,通过基于 C ++的操作符插件机制将其集成到 CuCIM 中。
Macenko 染色规范化和 CUDA C ++实现的 RGB2HED 函数都表现出显著的性能增益,与 NumPy 版本和纯 C ++版本相比,它们都表现出显著的性能增益。使用基于 NumPy 的过滤器,在 250 个图像数据集和 4000 × 4000 像素图像大小的情况下,训练 500 个时代以上的管道的染色归一化预处理时间大约为 13 天。 CuPy 基过滤器的过滤时间减少到 3 . 5 小时。
最终,加速数字病理学的前处理和后处理过滤器可以提高数字病理学深度学习管道的性能,加快数字病理学的采用,并使人工智能能够彻底改变病理学。