去年 11 月,一组来自 Alphabet ‘ sDeepmind 的计算生物学家利用人工智能从蛋白质的氨基酸序列中预测蛋白质的结构,解开了一个困扰科学家数十年的谜团。
甚至不到一年后,一项新的研究提供了一个更强大的模型,能够在一台游戏电脑上在短短 10 分钟内计算出蛋白质结构。
The research 来自华盛顿大学( UW )的科学家们承诺加快药物研发,这可以解开治疗癌症等疾病的方法。
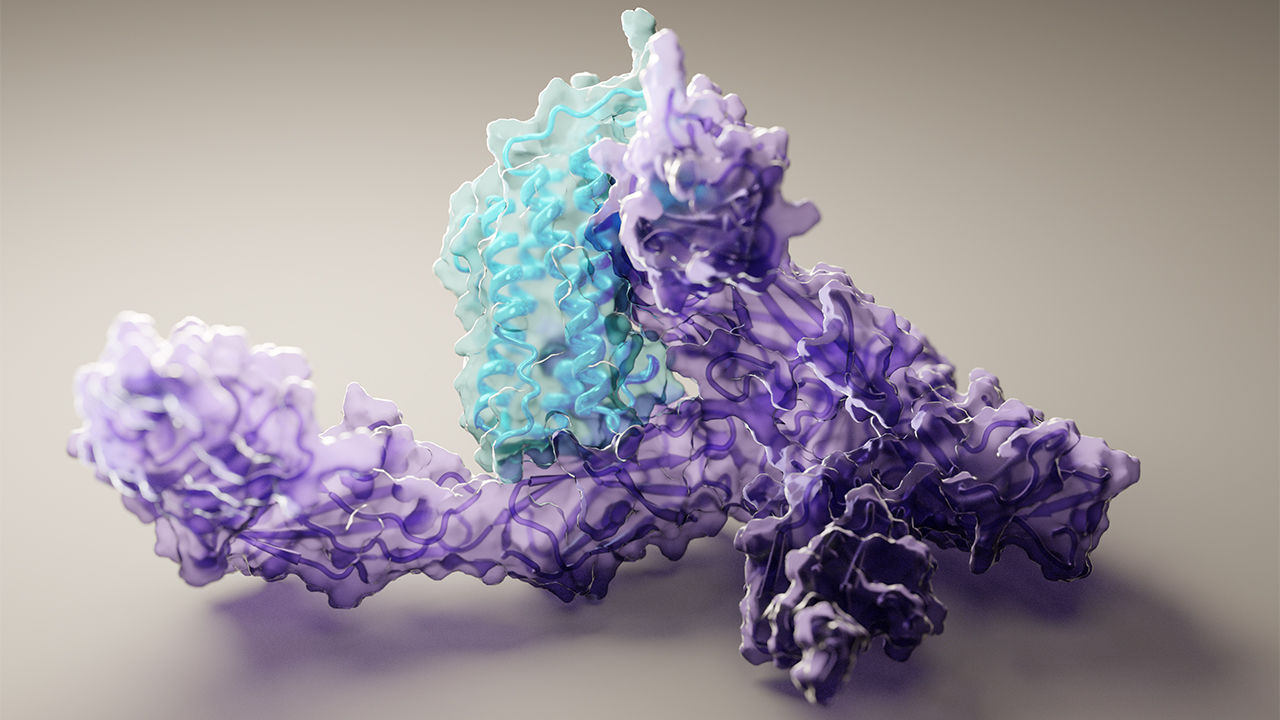
存在于身体的每一个细胞中,蛋白质在许多过程中发挥作用,如凝血、激素调节、免疫系统反应、视力以及细胞和组织修复。由长链氨基酸相互作用形成折叠的三维结构组成,蛋白质的形状决定其功能。
未折叠或错误折叠的蛋白质也被认为会导致退化性疾病,包括囊性纤维化、阿尔茨海默病、帕金森病和亨廷顿病。了解和预测一种蛋白质结构是如何形成的,可以帮助科学家为许多此类疾病设计有效的干预措施。
华盛顿大学的研究人员通过创建一个同时考虑蛋白质序列模式、氨基酸相互作用和可能的三维结构的三轨神经网络,开发了 RoseTTAFold 模型。
为了训练模型,研究小组使用不连续的蛋白质片段,有 260 个独特的氨基酸元素。具有the cuDNN – 加速 PyTorch 深度学习框架,以及 NVIDIA Geforce 2080 GPU ,这些信息在深度学习模型中来回流动。然后这个网络就可以推断出蛋白质的化学成分及其折叠结构。
“ RoseTTAFold 的端到端版本需要在 RTX 2080 GPU 上大约 10 分钟来生成少于 400 个残基的蛋白质的骨架坐标。研究人员在研究报告中写道:“ pyRosetta 版本需要 5 分钟对单个 NVIDIA RTX 2080 GPU 进行网络计算,用 15 个 CPU 核生成所有原子结构需要 1 小时。”。
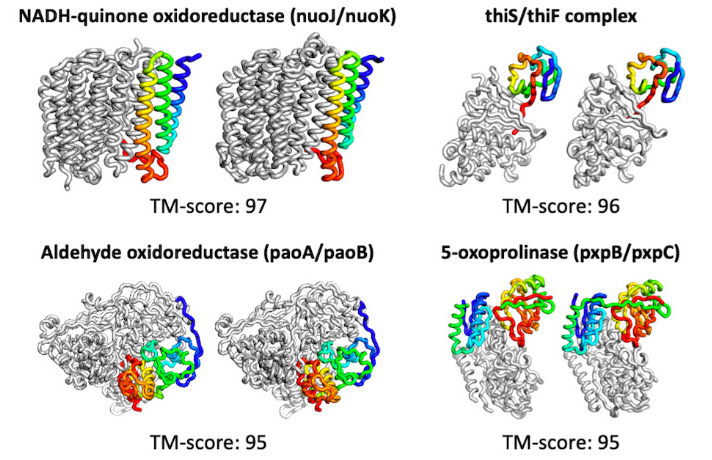
这个工具不仅可以快速预测蛋白质,而且可以在有限的输入下进行预测。它也有能力超越简单的结构进行计算,预测由几个结合在一起的蛋白质组成的复合物。更复杂的模型在 24G 上计算大约 30 分钟 NVIDIA Titan RTX .
任何有兴趣提交蛋白质序列的人都可以使用公共服务器。这个 源代码 科学界也可以免费获得。
“就在上个月,超过 4500 种蛋白质被提交到我们新的网络服务器上,我们通过 GitHub 网站提供了 RoseTTAFold 代码。我们希望这个新工具将继续有益于整个研究社区,”华盛顿大学蛋白质研究所的博士后学者 Minkyung Baek 说。