几十年来,分子模拟界在模拟势能面和原子间作用力时面临着精度与效率的两难选择。深势,人工神经网络力场,通过结合经典分子动力学( MD )模拟的速度和密度泛函理论( DFT )计算的准确性来解决这个问题。 1 这是通过使用 GPU – 优化包 DeePMD-kit 实现的,这是一个用于多体势能表示和 MD 模拟的深度学习包。 2
这篇文章提供了一个端到端的演示,演示如何为二维材料石墨烯训练神经网络潜力,并使用它在开源平台大型原子/分子大规模并行模拟器( LAMMPS )中驱动 MD 模拟。 3 培训数据可从维也纳从头算模拟软件包( VASP )获得 4 ,或量子浓缩咖啡( QE )。 5
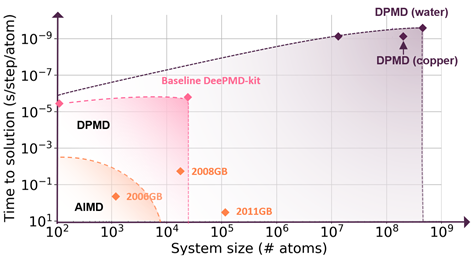
分子建模、机器学习和高性能计算( HPC )的无缝集成通过分子动力学和从头算准确性—这完全是通过基于容器的工作流来实现的。利用人工智能技术拟合 DFT 产生的原子间作用力,可以通过线性标度将可访问的时间和尺寸标度提高几个数量级。
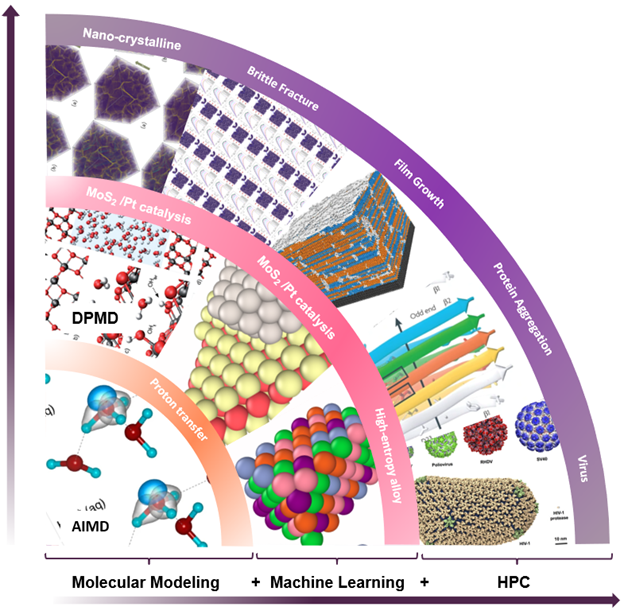
深度潜能本质上是机器学习和物理原理的结合,它开启了一种新的计算范式,如图 1 所示。
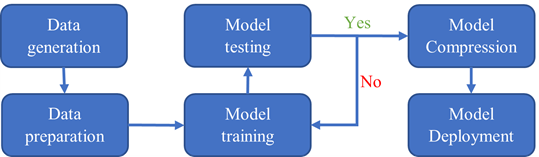
整个工作流如图 2 所示。数据生成步骤由 VASP 和 QE 完成。数据准备、模型训练、测试和压缩步骤使用 DeePMD 工具包完成。模型部署在 LAMMPS 中。
为什么是集装箱?
容器是一个可移植的软件单元,它将应用程序及其所有依赖项组合到一个与底层主机操作系统无关的包中。
本文中的工作流程涉及 AIMD 、 DP 培训和 LAMMPS MD 模拟。使用正确的编译器设置、 MPI 、 GPU 库和优化标志从源代码安装每个软件包是非常重要和耗时的。
容器通过为每个步骤提供一个高度优化的 GPU 支持的计算环境来解决这个问题,并且消除了安装和测试软件的时间。
NGC 目录是 GPU 优化的 HPC 和 AI 软件的集线器,它携带了整个 HPC 和 AI 容器 ,可以很容易地部署在任何 GPU 系统上。 NGC 目录中的 HPC 和 AI 容器经常更新,并进行可靠性和性能测试,这对于加快解决时间是必要的。
还将扫描这些容器的常见漏洞和暴露( CVE ),确保它们没有任何开放端口和恶意软件。此外, HPC 容器支持 Docker 和 Singularity 运行时,并且可以部署在云中或本地运行的多[ZFBB]和多节点系统上。
训练数据生成
模拟的第一步是数据生成。我们将向您展示如何使用 VASP 和 Quantum ESPRESSO 来运行 AIMD 模拟并为 DeePMD 生成训练数据集。可以使用以下命令从 GitHub 存储库下载所有输入文件:
git clone https://github.com/deepmodeling/SC21_DP_Tutorial.gitVASP
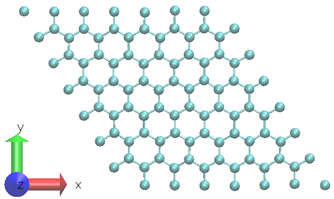
如图 3 所示,使用具有 98 个原子的二维石墨烯系统。 6 为了生成训练数据集,在 300K 下进行 0 . 5ps NVT AIMD 模拟。选择的时间步长为 0 . 5fs 。 DP 模型是使用固定温度下 0 . 5ps MD 轨迹的 1000 个时间步长创建的。
由于仿真时间较短,训练数据集包含连续的系统快照,这些快照高度相关。通常,训练数据集应从与各种系统条件和配置不相关的快照中采样。对于这个例子,我们使用了一个简化的训练数据方案。对于生产 DP 培训,建议使用 DP-GEN 利用并行学习方案,以有效探索更多的条件组合。 7
用投影增强波赝势描述了价电子与冻结核之间的相互作用。广义梯度近似交换− Perdew 的相关泛函−伯克−恩泽霍夫。在所有系统中,只有 Γ-point 用于 k-space 采样。
量子浓缩咖啡
AIMD 模拟也可以使用 Quantum ESPRESSO ( NGC 目录中的 container 提供)进行。 Quantum ESPRESSO 是一套基于密度泛函理论、平面波和赝势的开放源代码,用于 Nan oscale 的电子结构计算和材料建模。 QE 计算中使用了相同的石墨烯结构。以下命令可用于启动 AIMD 模拟:
$ singularity exec --nv docker://nvcr.io/hpc/quantum_espresso:qe-6.8 cp.x < c.md98.cp.in培训数据准备
一旦从 AIMD 仿真中获得训练数据,我们希望使用 dpdata 因此,它可以作为深层神经网络的输入。 dpdata 包是 AIMD 、 Classic MD 和 DeePMD 工具包之间的格式转换工具包。
您可以使用方便的工具 dpdata 将数据直接从 first principles 软件包的输出转换为 DeePMD 工具包格式。对于深势训练,必须提供物理系统的以下信息:原子类型、盒边界、坐标、力、病毒和系统能量。
快照或系统框架在一个时间步中包含所有原子的所有这些数据点,可以以两种格式存储,即 raw 和 npy 。
第一种格式 raw 是纯文本,所有信息都在一个文件中,文件的每一行表示一个快照。不同的系统信息存储在名为 box.raw, coord.raw, force.raw, energy.raw 和 virial.raw 的不同文件中。我们建议您在准备培训文件时遵循这些命名约定。
force.raw 的一个示例:
$ cat force.raw
-0.724 2.039 -0.951 0.841 -0.464 0.363 6.737 1.554 -5.587 -2.803 0.062 2.222
-1.968 -0.163 1.020 -0.225 -0.789 0.343这个 force.raw 包含三个框架,每个框架具有两个原子的力,形成三条线和六列。每条线在一帧中提供两个原子的所有三个力分量。前三个数字是第一个原子的三个力分量,而下三个数字是第二个原子的力分量。
坐标文件 coord.raw 的组织方式类似。在 box.raw 中,应在每行上提供盒向量的九个分量。在 virial.raw 中,维里张量的九个分量应按 XX XY XZ YX YY YZ ZX ZY ZZ 的顺序提供在每一行上。所有原始文件的行数应相同。我们假设原子类型不会在所有帧中改变。它由 type.raw 提供,它有一行原子类型,一行一行地写。
原子类型应该是整数。例如,一个系统的 type.raw 有两个原子,分别为零和一:
$ cat type.raw
0 1将数据格式转换为 raw 不是一项要求,但此过程应能说明可作为培训用 DeePMD 工具包输入的数据类型。
将第一原理结果转换为训练数据的最简单方法是将其保存为 NumPy 二进制数据。
对于 VASP 输出,我们准备了一个 outcartodata.py 脚本来处理 VASP OUTCAR 文件。通过运行以下命令:
$ cd SC21_DP_Tutorial/AIMD/VASP/
$ singularity exec --nv docker://nvcr.io/hpc/deepmd-kit:v2.0.3 python outcartodata.py
$ mv deepmd_data ../../DP/量化宽松产出:
$ cd SC21_DP_Tutorial/AIMD/QE/
$ singularity exec --nv docker://nvcr.io/hpc/deepmd-kit:v2.0.3 python logtodata.py
$ mv deepmd_data ../../DP/生成名为 deepmd_data 的文件夹并将其移动到培训目录。它生成五组 0/set.000, 1/set.000, 2/set.000, 3/set.000, 4/set.000 ,每组包含 200 帧。不需要处理每个 set .*目录中的二进制数据文件。包含 set.* 文件夹和 type.raw 文件的路径称为系统。如果要训练非周期系统,应在系统目录下放置一个空 nopbc 文件。 box.raw 不是必需的,因为它是非周期系统。
我们将使用五套中的三套进行培训,一套用于验证,另一套用于测试。
深势模型训练
深势模型的输入是包含前面提到的系统信息的描述符向量。神经网络包含几个隐藏层,由线性和非线性变换组成。在这篇文章中,使用了一个三层神经网络,每层有 25 个、 50 个和 100 个神经元。神经网络学习的目标值或标签是原子能。训练过程通过最小化损失函数来优化权重和偏差向量。
训练由命令启动,其中 input.json 包含训练参数:
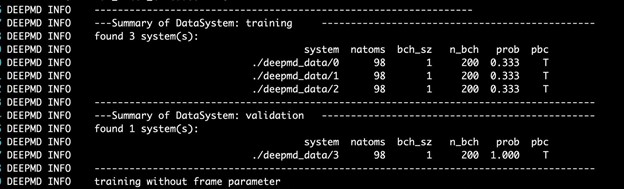
$ singularity exec --nv docker://nvcr.io/hpc/deepmd-kit:v2.0.3 dp train input.jsonDeePMD 工具包打印培训和验证数据集的详细信息。数据集由输入脚本的 training 部分中定义的 training_data 和 validation_data 确定。训练数据集由三个数据系统组成,而验证数据集由一个数据系统组成。原子数、批次大小、系统中的批次数以及使用系统的概率均如图 4 所示。最后一列显示系统是否假设周期边界条件。
在培训期间,每 disp_freq 培训步骤都会使用用于培训模型的批次和验证数据中的 numb_btch 批次测试模型的错误。在文件 disp_file 中相应地打印训练错误和验证错误(默认为 lcurve.out )。可在输入脚本中通过训练和验证数据集相应部分中的键 batch_size 设置批量大小。
输出的一个示例:
# step rmse_val rmse_trn rmse_e_val rmse_e_trn rmse_f_val rmse_f_trn lr 0 3.33e+01 3.41e+01 1.03e+01 1.03e+01 8.39e-01 8.72e-01 1.0e-03 100 2.57e+01 2.56e+01 1.87e+00 1.88e+00 8.03e-01 8.02e-01 1.0e-03 200 2.45e+01 2.56e+01 2.26e-01 2.21e-01 7.73e-01 8.10e-01 1.0e-03 300 1.62e+01 1.66e+01 5.01e-02 4.46e-02 5.11e-01 5.26e-01 1.0e-03 400 1.36e+01 1.32e+01 1.07e-02 2.07e-03 4.29e-01 4.19e-01 1.0e-03 500 1.07e+01 1.05e+01 2.45e-03 4.11e-03 3.38e-01 3.31e-01 1.0e-03
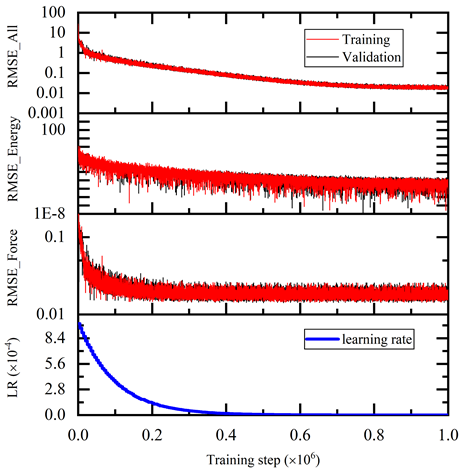
如图 5 所示,训练误差随着训练步骤单调减少。训练后的模型在测试数据集上进行了测试,并与 AIMD 仿真结果进行了比较。测试命令是:
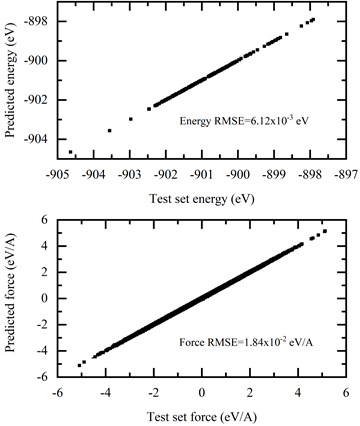
$ singularity exec --nv docker://nvcr.io/hpc/deepmd-kit:v2.0.3 dp test -m frozen_model.pb -s deepmd_data/4/ -n 200 -d detail.out结果如图 6 所示。
模型导出和压缩
模型训练完成后,生成一个冻结模型,用于 MD 仿真中的推理。从检查点保存神经网络的过程称为“冻结”模型:
$ singularity exec --nv docker://nvcr.io/hpc/deepmd-kit:v2.0.3 dp freeze -o graphene.pb生成冻结模型后,可以在不牺牲精度的情况下对模型进行压缩;在 MD 中大大加快推理性能的同时,根据仿真和训练设置,模型压缩可以将性能提高 10 倍,在 GPU 上运行时将内存消耗减少 20 倍。
可以使用以下命令压缩冻结模型, -i 表示冻结模型, -o 表示压缩模型的输出名称:
$ singularity exec --nv docker://nvcr.io/hpc/deepmd-kit:v2.0.3 dp compress -i graphene.pb -o graphene-compress.pbLAMMPS 中的模型部署
在 LAMMPS 中实现了一种新的配对方式,以便在前面的步骤中部署经过训练的神经网络。对于熟悉 LAMMPS 工作流程的用户,只需进行最小的更改即可切换到深度潜力。例如,具有 Tersoff 电位的传统 LAMMPS 输入具有以下电位设置:
pair_style tersoff
pair_coeff * * BNC.tersoff C若要使用深电位,请将以前的线路替换为:
pair_style deepmd graphene-compress.pb
pair_coeff * *输入文件中的 pair_style 命令使用 DeePMD 模型来描述石墨烯系统中的原子相互作用。
graphene-compress.pb文件表示用于推断的冻结和压缩模型。- MD 模拟中的石墨烯系统包含 1560 个原子。
- 周期性边界条件应用于横向
x和y方向,自由边界应用于z方向。 - 时间步长设置为 1 fs 。
- 将系统置于温度为 300 K 的 NVT 系综下进行松弛,这与 AIMD 设置一致。
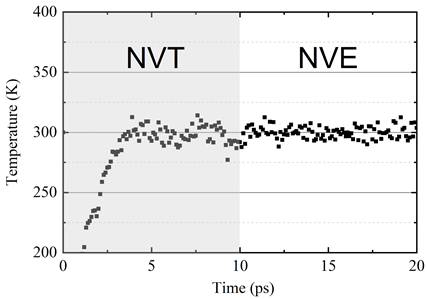
NVT 松弛后的系统配置如图 7 所示。可以观察到,深势可以描述原子结构,在横平面方向上有小的波纹。在 10ps NVT 松弛后,将系统置于 NVE 系综下以检查系统稳定性。
系统温度如图 8 所示。
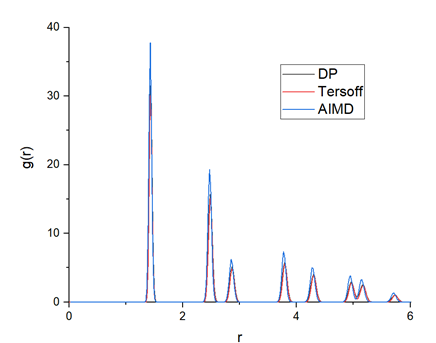
为了验证经过训练的 DP 模型的准确性,从 AIMD 、 DP 和 Tersoff 计算出的径向分布函数( RDF )如图 9 所示。 DP 模型生成的 RDF 与 AIMD 模型非常接近,这表明 DP 模型可以很好地描述石墨烯的晶体结构。
结论
这篇文章展示了在给定条件下石墨烯的一个简单案例研究。 DeePMD-kit 软件包简化了从 AIMD 到经典 MD 的工作流程,具有很大的潜力,提供了以下关键优势:
- 在 TensorFlow 框架中实现高度自动化和高效的工作流。
- 使用流行的 DFT 和 MD 包(如 VASP 、 QE 和 LAMMPS )的 API 。
- 广泛应用于有机分子、金属、半导体、绝缘体等。
- 具有 MPI 和[ZFBB]支持的高效 HPC 代码。
- 模块化,便于其他深度学习潜在模型采用。
此外,使用 NGC 目录中的 GPU – 优化容器简化并加速了整个工作流程,省去了安装和配置软件的步骤。要为其他应用程序培训综合模型,请从 NGC 目录下载 DeepMD Kit Container 。
致谢
我们感谢与坦普尔大学的张春义博士、韩丹博士、山东大学的王新宇博士以及 DeepModeling 社区的张林峰博士、张玉芝博士、曾金哲博士、张铎博士和袁凤波博士进行的有益讨论。
工具书类
[1] 贾伟,王浩,陈明,陆德,林力,车 R , E W 和张力 2020 年推动了分子动力学的极限从头算机器学习精确到 1 亿个原子 IEEE 出版社 5 1-14
[2] Wang H , Zhang L , Han J 和 E W 2018 DeePMD 工具包:多体势能表征和分子动力学计算机物理通信的深度学习包 228 178-84
[3] Plimpton S 1995 短程分子动力学快速并行算法计算物理杂志 117 1-19
[4] Kresse G 和 Hafner J 1993 液态金属的从头算分子动力学物理评论 B 47 558-61
[5] 吉安诺齐 P 、巴罗尼 S 、博尼尼 N 、卡兰德拉 M 、卡尔 R 、卡瓦佐尼 C 、塞雷索尔 D 、基亚罗蒂 G L 、科科科西科尼 M 、达博 I 、达尔科索 A 、德吉隆科利 S 、法布里斯 S 、弗拉迪斯 G 、格鲍尔 R 、格斯特曼 U 、古古古西 C 、科卡利 A 、拉泽里 M 、马丁萨莫斯 L 、马扎里 N 、毛里 F 、马扎雷罗 R 、保利尼 S 、帕斯夸雷罗 A 、保拉托 L 、斯巴克亚 C 、斯堪多洛 S 、, Sclauzero G 、 Seitsonen A P 、 Smogunov A 、 Umari P 和 Wentzcovitch R M 2009 量子浓缩咖啡:材料量子模拟的模块化开源软件项目物理学杂志:凝聚态 21 395502
[6] Humphrey W , Dalke A 和 Schulten K 1996 VMD :分子图形可视化分子动力学杂志 14 33-8
[7] 张玉芝、王海迪、陈伟杰、曾金哲、张林峰、王汉和魏 Nan E , DP-GEN :基于可靠深度学习的势能模型生成的并行学习平台,计算机物理通信, 2020 , 107206 。